 Molexus 社 Website へ
Northern Science Consulting
Molexus 社 Website へ
Northern Science Consulting
Molegro Virtual Docker は、リガンド-蛋白質相互作用を予測するためにMolDockドッキングエンジンを用いています。MolDockは、guided differential evolution と呼ばれる新しいハイブリッドサーチアルゴリズムをベースにしています。
Guided differential evolution アルゴリズムは、ドッキングプロセスの実行中に動的に用いられるキャビティ予測アルゴリズムに、differential evolution (DE) 最適化手法を組み合わせたものです。
Differential evolutionは、1995年にStornとPrice[1]によって紹介され、これまで様々な最適化問題に適用され成功を収めてきました。サーチプロセスが行われている間の、予測キャビティの使用は、高速かつ高精度なポテンシャルバインディングモードの同定を可能にします。
ドッキングシミュレーションで用いられる、MolDockスコアリング関数は、区分線形ポテンシャル = "piecewise linear potential" (PLP) に基づいています[3]。MolDockでは、ドッキングスコアリング関数が、水素結合指向性を考慮した項と新しいチャージスキームよって拡張されています。このドッキングスコアリング関数により、ドッキングプロセス中にposeを瞬時に評価することが可能となりました。
ドッキングスコアリング関数、 Escore は、次のエネルギー項によって定義されます:

Einter は、リガンド-タンパク質相互作用エネルギーで:
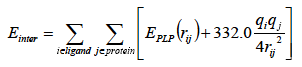
この中で、EPLP 項は区分線形ポテンシャルであり、第2項は、帯電原子間の静電相互作用を示しており、
Eintra は、リガンドの内部エネルギーで:
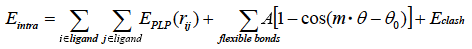
ドッキングプロセスの終了後、得られたポーズは、更に複雑な力場(LJ12-6を用いた項)を用いて再びランク付けすることも可能です。
更に詳しい情報については、Molegro Virtual Dockerのマニュアルをご参照ください。MolDockスコアリング関数と検索アルゴリズムに関する詳細な解説が掲載されています。
[1] Storn, R.; Price, K. Differential Evolution - A Simple And Efficient Adaptive Scheme for Global Optimization over Continuous Spaces. Tech-report, International Computer Science Institute, Berkley, 1995.
[2] Friesner, R. A.; Banks, J. L.; Murphy, R. B.; Halgren, T. A. Glide: A New Approach for Rapid Accurate Docking And Scoring. 1. Method And Assessment of Docking Accuracy. J. Med. Chem. 2004, 47, 1739-1749.
[3] Gehlhaar, D. K.; Verkhivker, G.; Rejto, P. A.; Fogel, D. B.; Fogel, L. J.; Freer, S. T. Docking Conformationally Flexible Small Molecules Into a Protein Binding Site Through Evolutionary Programming. Proceedings of the Fourth International Conference on Evolutionary Programming 1995, 615-627.
©2004-2025 Northern Science Consulting Inc. All Rights Reserved.