 Molexus 社 Website へ
Northern Science Consulting
Molexus 社 Website へ
Northern Science Consulting
分子のインポートとプレパレーションは非常に容易です。分子構造ファイルは、その構成要素別(タンパク質、リガンド、補因子=cofactor、水分子)に取り扱われ、自動的にプレパレートされます。
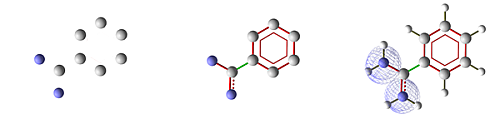
MVDが、結合、芳香族性を検出し、チャージをアサインします。そして、explicitな水素を付加します。
ビルトインされたキャビティ検出機能が、有望なバインディング位置を見つけ出します。最も興味のある領域に対してサーチ空間を制限することも可能です。
プロトン化ウィザードにより、可能性のあるプロトン化ターゲット(例えば、ヒスチジン残基)を迅速に同定し、コンテキストメニューを用いてすばやくそれらの水素化状態を変更することができます。
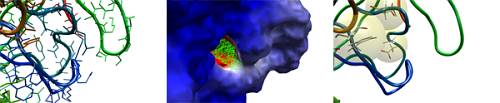
自動キャビティ検出とプロトン化ガイドを用いたプロテインのプロトン化
ドッキングプロセスは、ドッキングウィザードを通して行われます。ドッキングウィザードにより次のような操作が可能です:
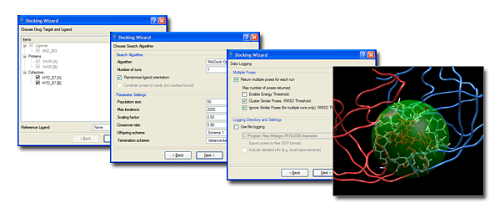
Docking Wizard でのいろいろなステップ
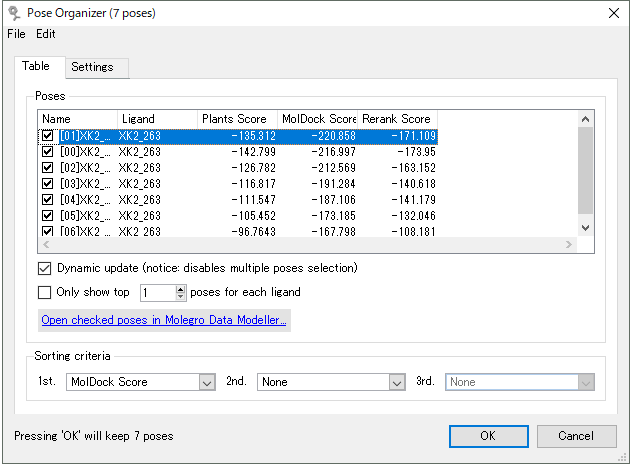
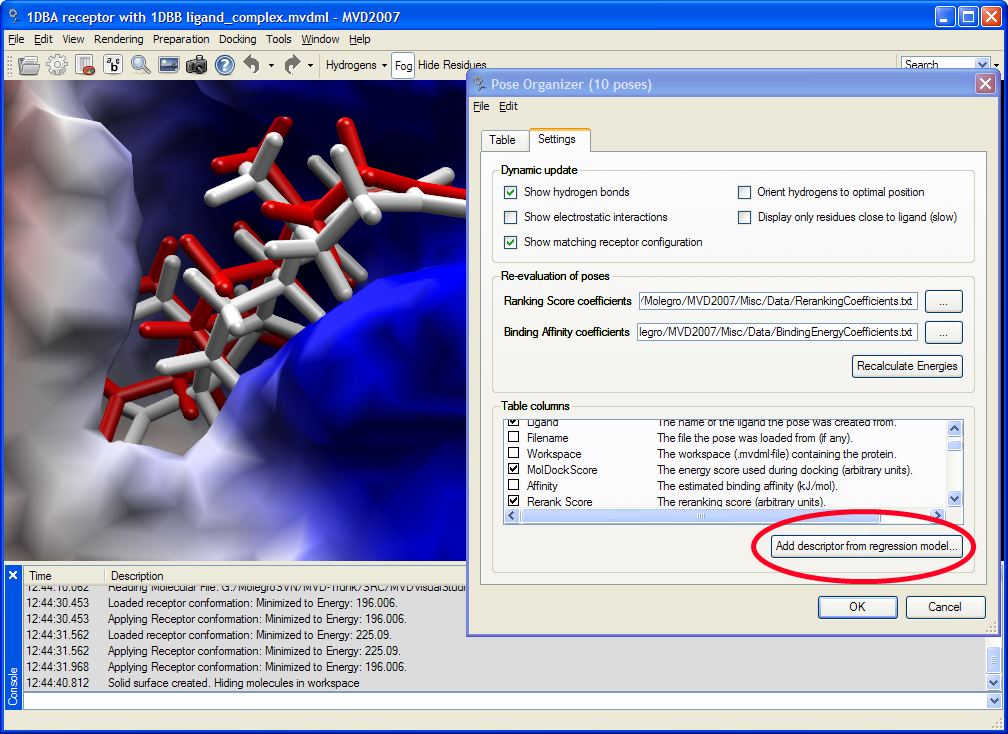
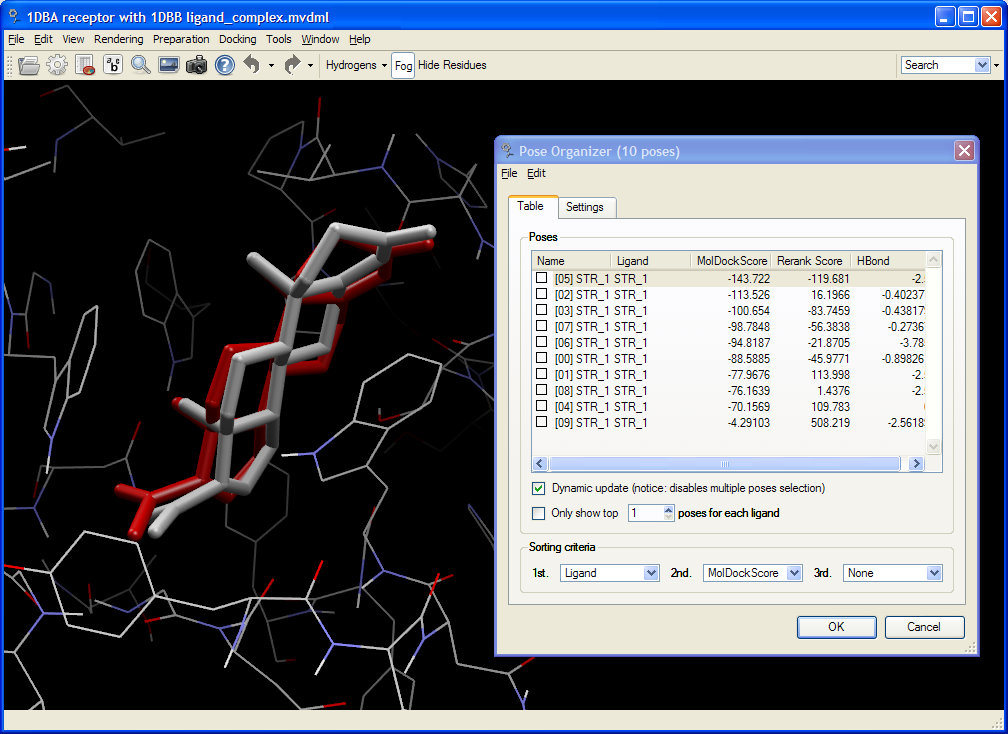
Pose Organizer で、ドッキングエンジンによって返されたポーズをブラウズすることができます。ドッキングを実行しながらポーズをロードすることができるので、非常に多くのリガンドをブラウジングすることを可能にしています。
また、様々なエネルギー項と相互作用を検証することができることに加え、更に高度な順位付けとバインディングアフィニティ量を計算することも可能です。ポーズが切り替わるとき、水素結合と静電相互作用は、それに合わせて更新されます。
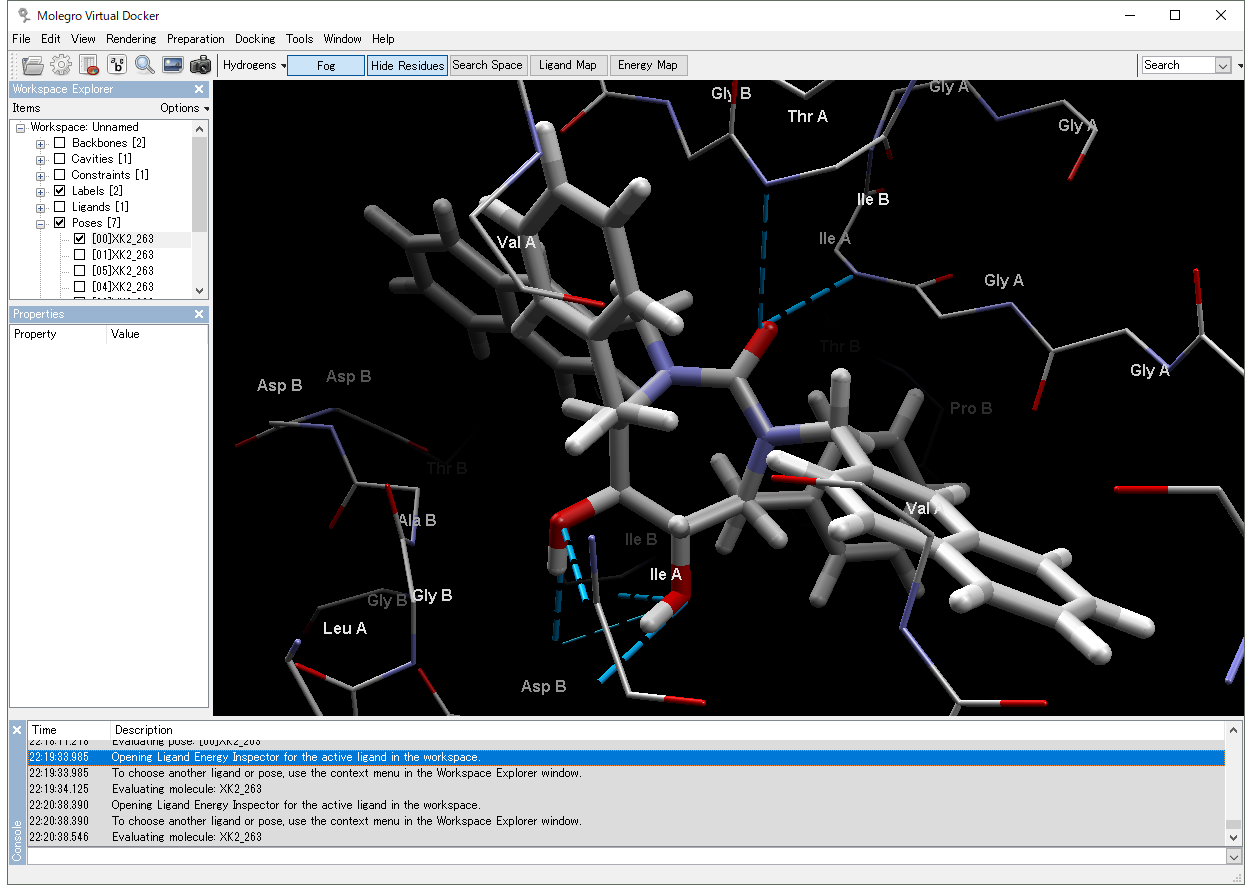
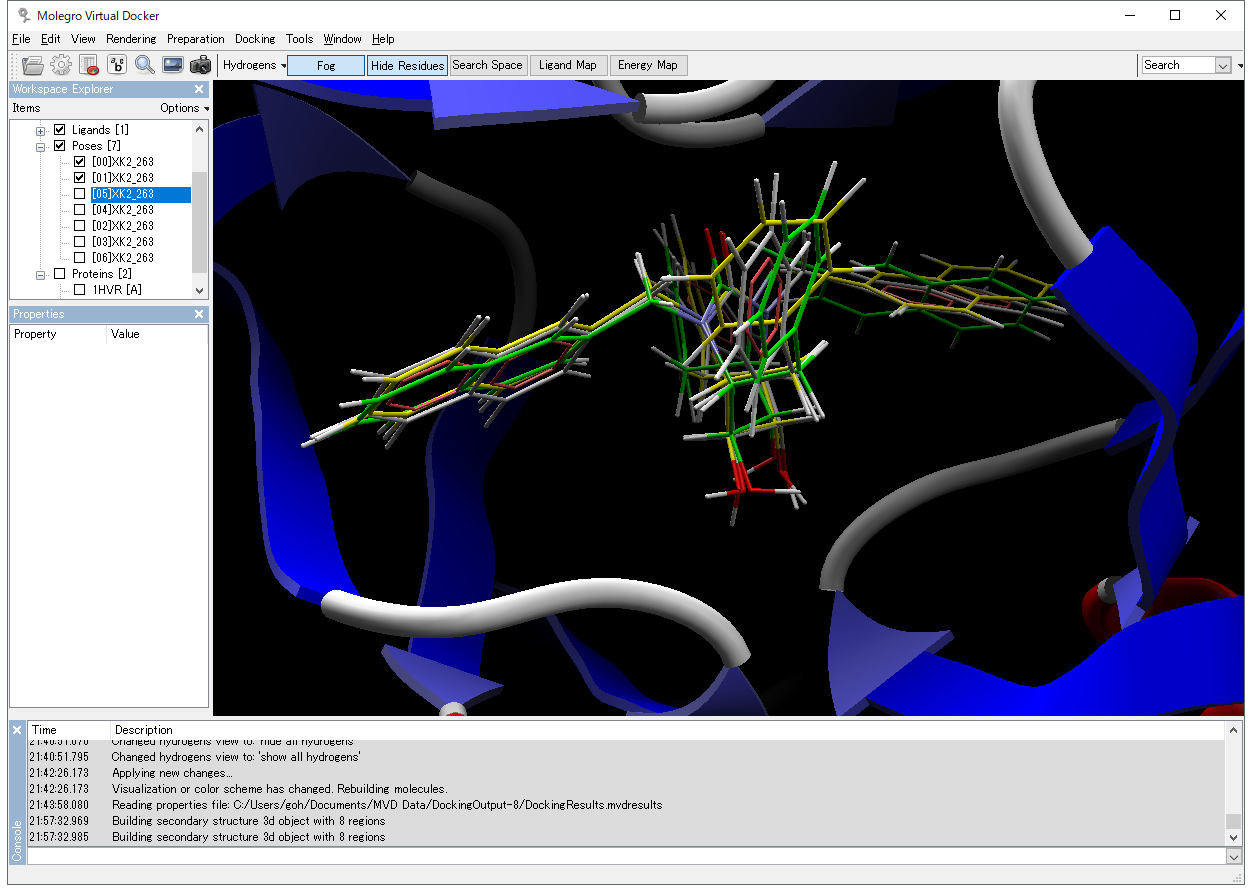
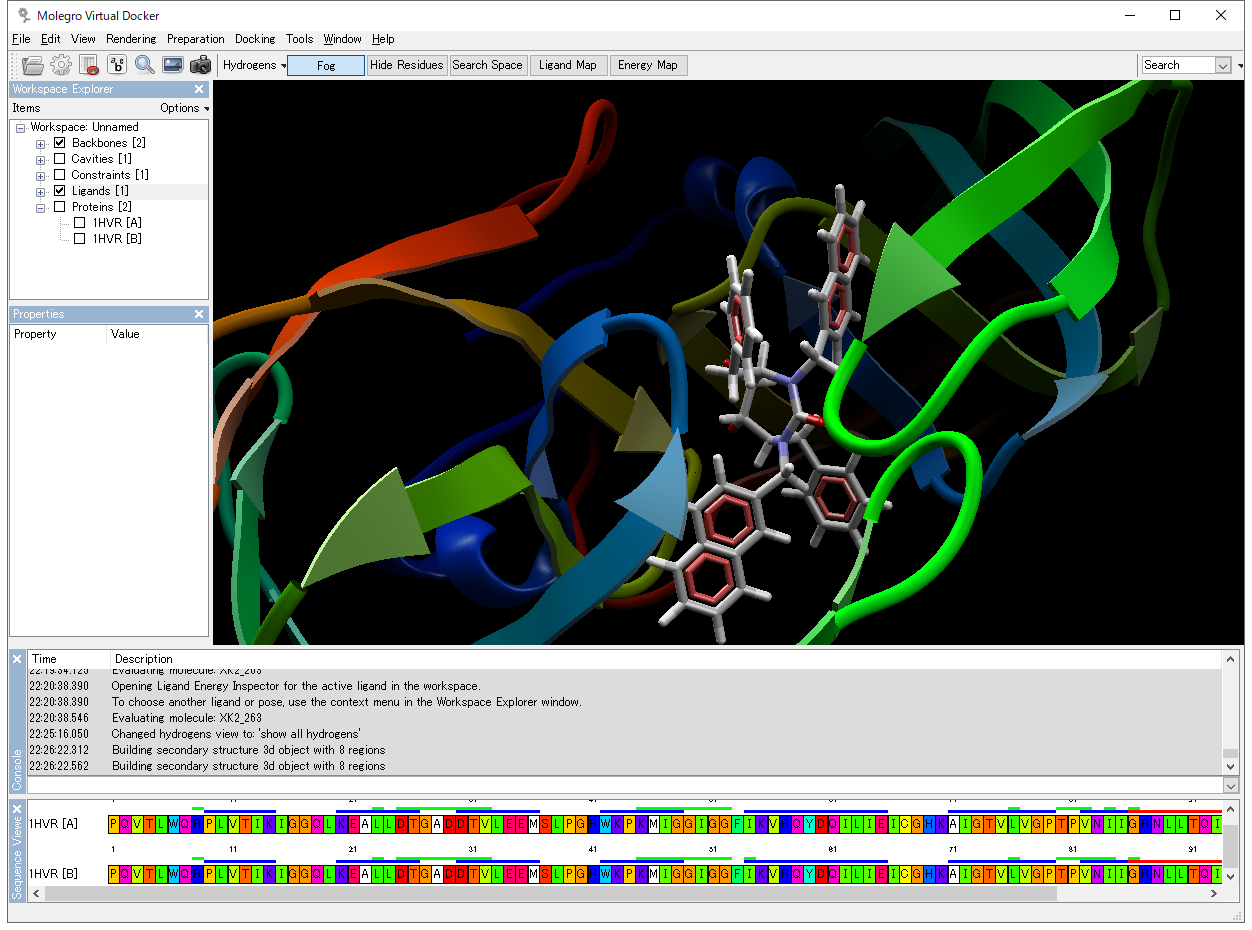
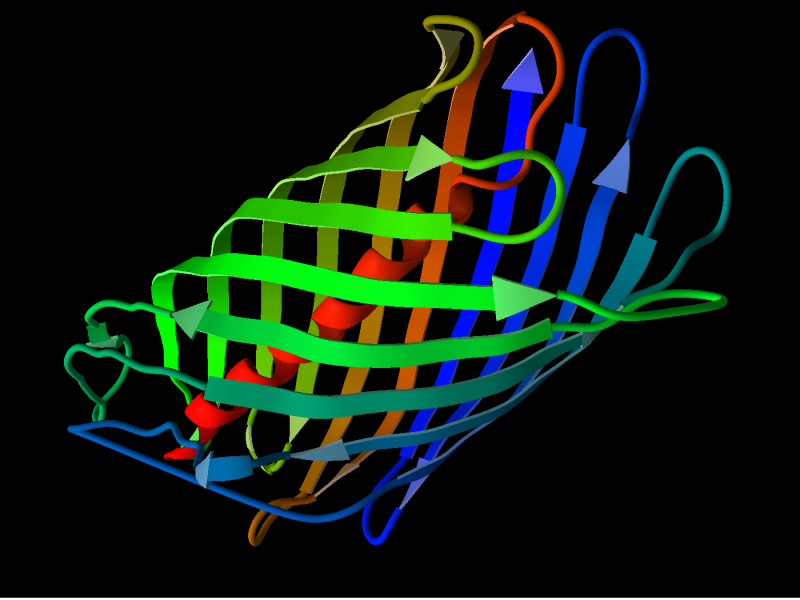
Sequence Viewer では、蛋白質中の残基を素早く識別し選択することができます。カートーン表示では、異なる二次構造領域を強調表示します。
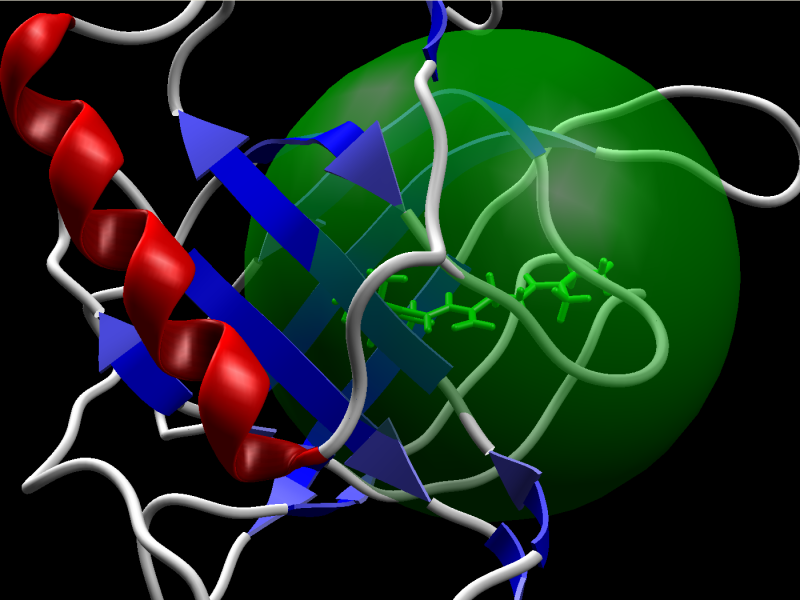
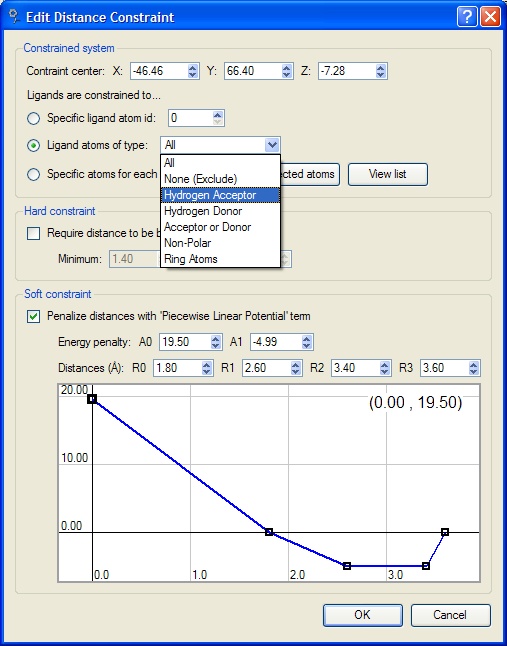
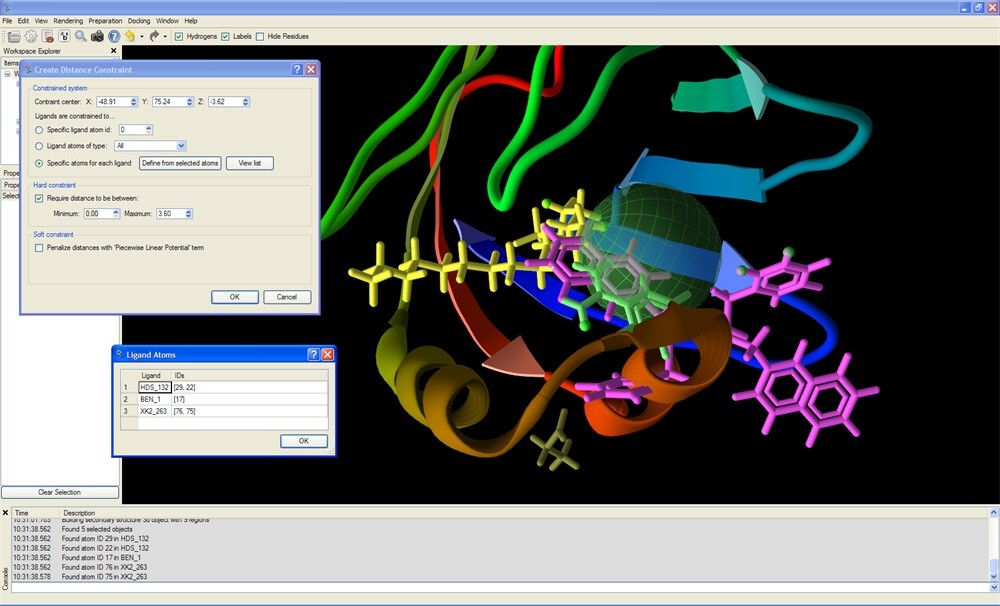
様々な束縛により、エネルギー景観の変更、相互作用発生の強制や抑制が可能です。
束縛は、化学特性(例えば、水素結合受容体や環原子)に基づいた定義、あるいは、各リガンドの個々の原子ついて指定が可能です。
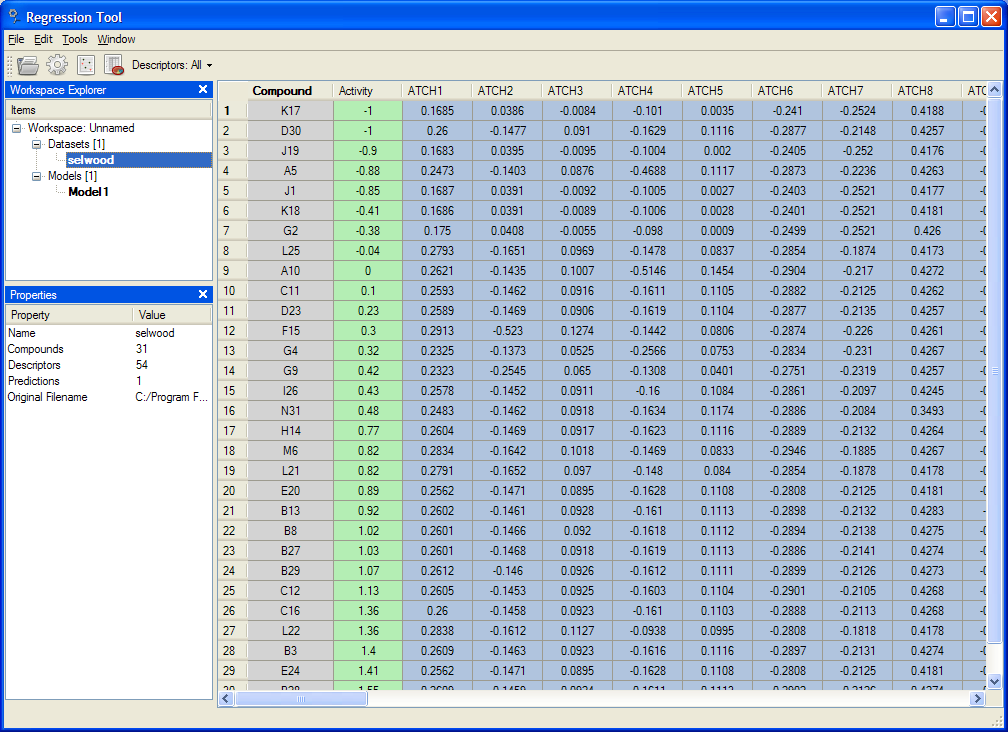
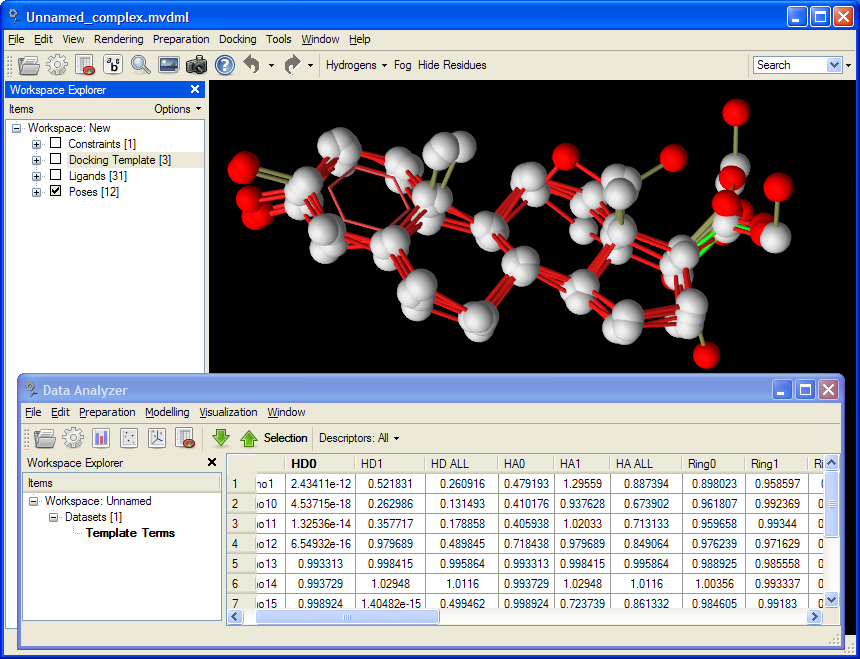
MVD にビルトインされている データモデラーは、ニューラルネットや重線形回帰の回帰モデルの生成、統計量の抽出、高次元データを含むデータの可視化を行います。
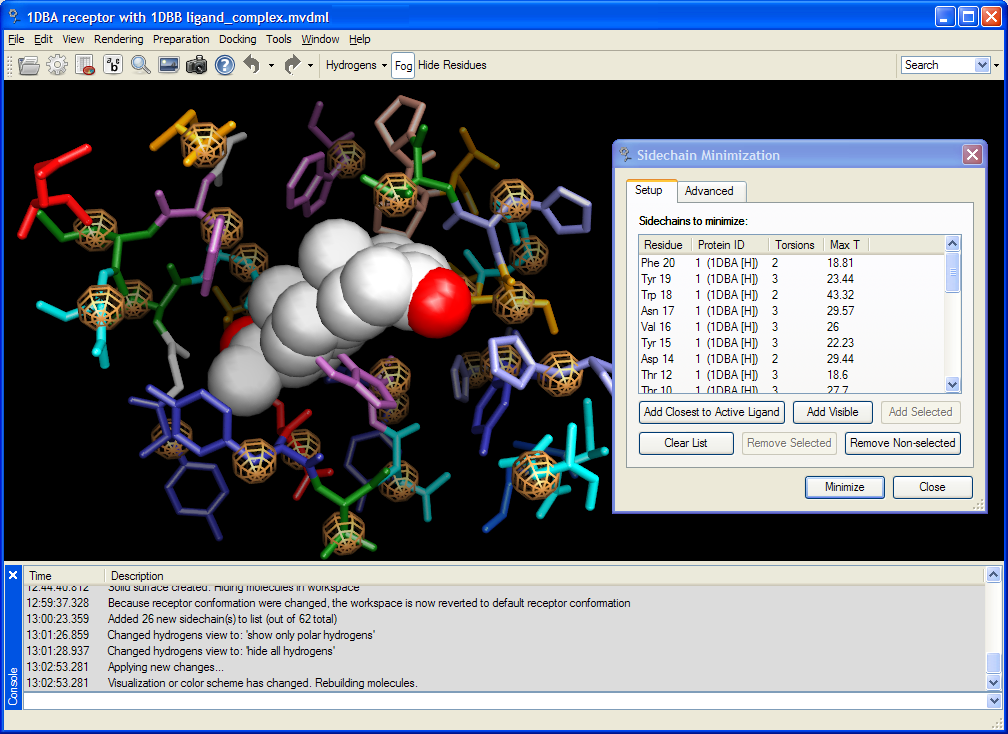
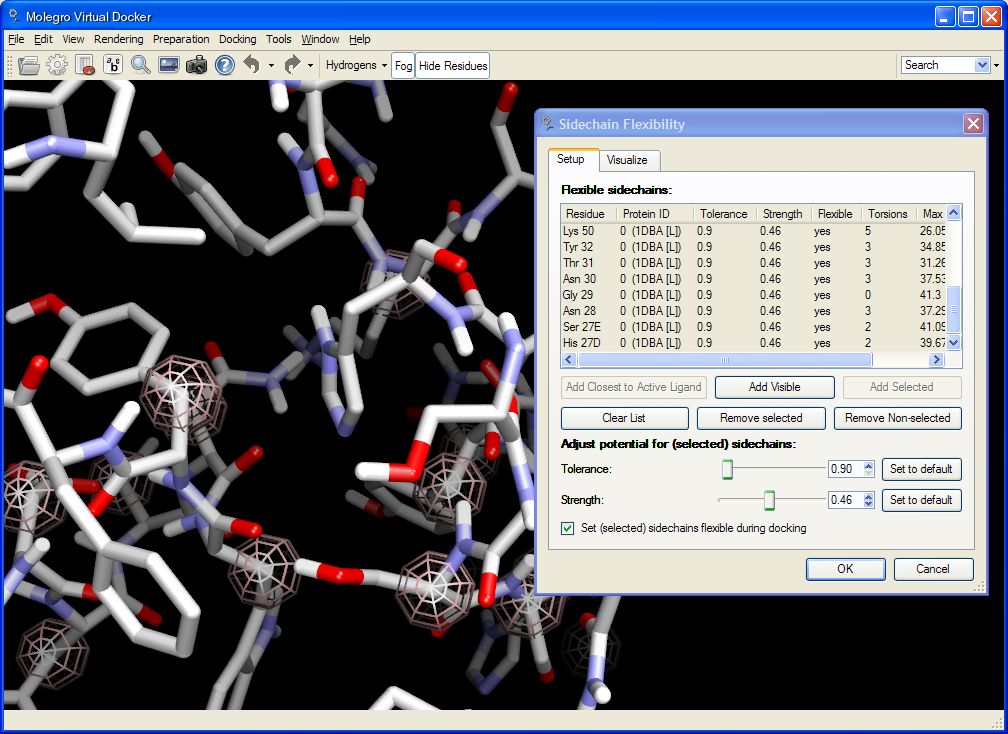
MVDでは、サイドチェーンフレキシビリティは、ドッキング計算時のポテンシャルをソフトにし(PLP-ポテンシャルのトレランスを増加させる、あるいは、選択されたサイドチェーン相互作用を弱める)、多様なポーズのセットをドッキングし、そして、最終的に側鎖の構造を最適化することで行われます。レセプター構造を手動で最小化することも可能です。
ドッキング計算が完了した後、見つかったポーズは、それらのマッチングレセプターコンフォメーションと同時にポーズオーガナイザーで直接視覚的に検証できます。
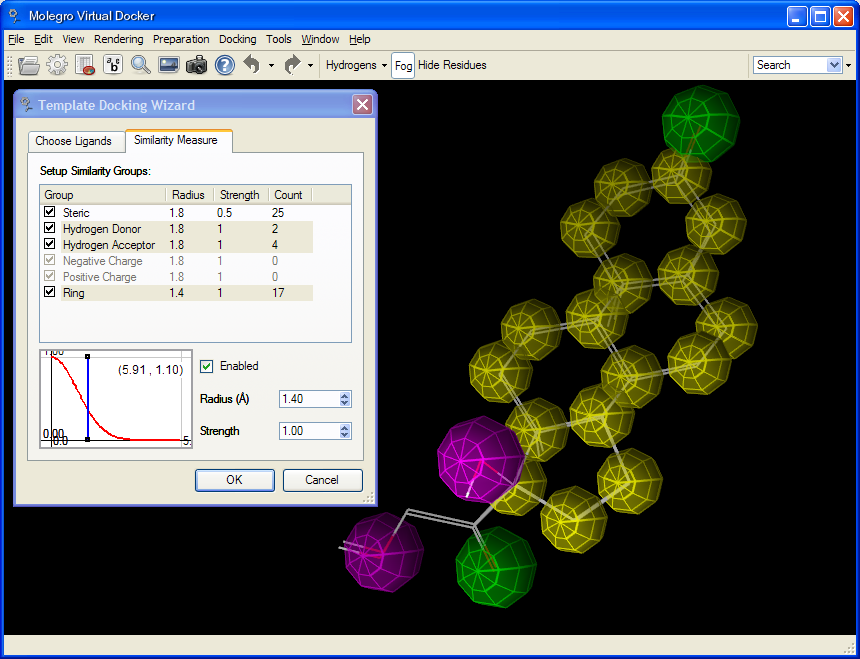
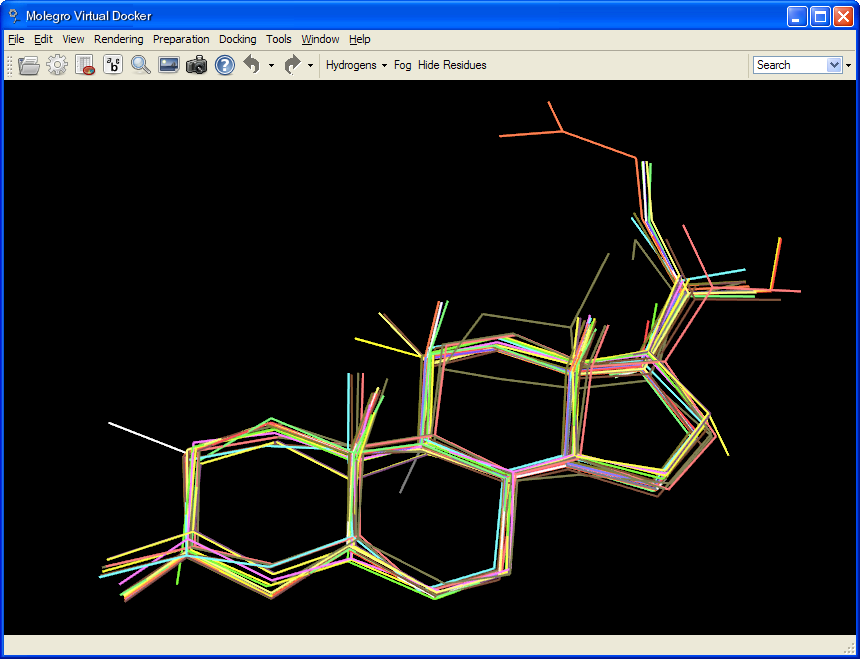
MVDでは、リガンドをドッキングテンプレートに対してドックさせることが可能です。ファーマコフォーと同じように、テンプレートは関連性のある化学プロパティ(チャージ、水素結合性のような)から構築され、一つあるいは複数のリガンドコンフォメーションから自動生成させることができます。 テンプレートはいろいろは方法で利用することができます:
©2004-2025 Northern Science Consulting Inc. All Rights Reserved.