ノーザンサイエンスコンサルティング株式会社
ノーザンサイエンスコンサルティング株式会社

ADMET Predictor® は、化合物構造から ADMET 物性を高速・高精度に予測します。第三者による数多くの論文で最も精度が高いとの評価を得ています。また、ユーザーデータに基づく独自の予測モデルを構築するモジュールも準備されており、in silico 初心者からエキスパートまで幅広くご利用いただけます。
2025年6月4日に、新バージョン ADMET Predictor® 13 の日本向けリリースウェビナーが開催されました。ウェビナービデオでご確認ください。
このバージョンから、ライセンスシステムが Flexera から Reprise (リプリーズ) に変更されました。 ADMETPredictor 13 を使用するには、Reprise ライセンスサーバーおよび Reprise 用のライセンスが必要となります。Flexera から Reprise への移行方法についてはこちらのページを参照してください。
HTPK モジュールでは以下の機能強化が施されました:
以上
このバージョンには、新しい予測モデルとして、肝細胞非結合分画 (S+fuhep)、PAMPA 透過性 (S+PAMPA)、および CYP1A2 誘導 (CYP1A2_Ind) が加わりました。また、低排出 MDCK 透過性 (S+MDCK-LE) の新しい回帰モデルもあり、これは同じ名前の以前の分類モデルに代わるものです。
新しいデータの追加により、次のモデルが改良されました。
薬物誘発性肝障害(DILI)を予測する定量的システム毒性学ソフトウェア DILIsym へのインプットに焦点を当てた新しいモジュールが搭載されました。このモジュールには、ミトコンドリア機能不全、活性酸素種(ROS)毒性、MRP3(多剤耐性関連タンパク質3)、BSEP(胆汁酸塩排出ポンプ)、MDR3(多剤耐性タンパク質3)トランスポーターの阻害を予測する 11 のモデルが含まれています。今回のリリースでは、BSEP モデル(BSEP_Inh および BSEP_IC50)以外、すべて新たに加わったモデルです。
DILIsym モジュールには、AP_DILISYM ライセンスが必要となりますが、前述の 2 つの BSEP モデルについては、AP_TRANSPORTERS ライセンスで使用することができます。
HTPK モジュールに、以下の機能強化が加えられました。
このリリースには、Python エコシステム内で ADMET Predictor の機能にアクセスするための pyADMETPredictor というモジュールが含まれています。このモジュールは、ADMET Predictor コマンドライン、または REST API のいずれかで利用できます。リリースには、Jupyter ノートブックが含まれており、いくつかの例を用いてモジュールの使用方法を説明しています。
このバージョンでは、数値重みを使用して各目的変数の相対的重要度を指定する機能が提供されています。このような重みが指定される場合、仮想化合物のランク付けと選択は、パレート最適化ではなく、多基準決定分析 (MCDA) に基づいて行われます。
HTPK モジュール内で計算された新しい組織分配係数に基づいて、14 種類の異なる組織への選択性を最適化するための新しい目的変数 <TissueSelectivity_*> が利用可能になりました。
参照構造との 3D 類似性に基づく目的変数(3D Similarity)が AIDD のメイン画面から利用できるようになりました。個別のパラメーターファイルは不要になりました。
また、分子変換のデータベースにいくつかの機能強化が行われました。
「線形ブーストニューラルネットワーク」と呼ばれる新しいモデリング手法が追加されました。これは従来のニューラルネットワークの拡張であり、入力層と出力層を直接接続する追加の重みセットを組み込んだものです。この手法は、従来のニューラルネットワークの出力が従属変数に対して圧縮された範囲を持つ場合に特に役立ちます。これは、特にS/N 比の低い小規模なデータセットでよく見られる現象です。
35 を超える新しいモデリングディスクリプターが追加されています。
ADMET Predictor の “pKa Microstates Display” の画像ファイルとチャートデータを /predict_admet または /run_cmd URI を使用して生成できるようになりました。
ADMET Predictor サービスが起動時に一度だけライセンスをチェックアウトするのではなく、リクエストごとにライセンスをチェックアウトする場合に使用する、新しいオプションの JSON キー "licenses" が追加されました。このキーが指定されると、サービスは指定されたライセンスの即座のチェックアウトを試み、ライセンスが利用できない場合は戻ります。これにより、そのような事象の検出が容易になります。
MedChem Designer は次のように機能が強化されました。
スクリプトファイルワークフロー (ms_generate_pKa_images.txt) が新たに追加されました。これは、”pKa Microstates Display” から画像ファイルとチャートデータを生成します。
新しいコマンドライン引数 -species を使用すると、ユーザーは HTPK シミュレーションの species を指定された種だけに制限することができます。
3D形状マッチングは、既存の3D座標が使用されている場合、切断されたフラグメントに対応するようになりました。
以下の不具合を修正しました:
以上
更新・新機能については、ADMET Predictor® 13.0 のリリースノート: ADMET Predictor® 13.0 - Jul 25, 2025 でご確認ください。
以上
更新・新機能については、ADMET Predictor® 12.0 のリリースノート: ADMET Predictor® 12.0 - Aug 6, 2024 でご確認ください。
以上
更新・新機能については、以下にある、ADMET Predictor® 11.0 のリリースノート: ADMET Predictor® 11.0 - July 18, 2023 でご確認ください。
以上
実験から得たイオン化定数の大規模なデータセットでの入念な再トレーニングにより、pKaモデルが大幅に改善されました。このデータセットには、パートナー企業 3 社から提供された、それぞれ、約 19,000 化合物、2,400 化合物、4,100 化合物という独自データが組み込まれており、全体的なイオン化定数の数は、バージョン 10.4 の 33,640 から、本バージョンでは 70,810 に増加しています。モデルの適用領域の拡大により、Simulations Plus 社およびパートナー企業からの外部テストセットの大部分において、より一層正確な予測が可能になりました。
予測 pK50 値の表示が改善されました。ユーザーの利便性のために、Detailed Info feature of Ionization Graphs にそれらが追加されました。さらに、カメレオン性基の場合、基ごとに複数のpK50 の存在が了承され、pK50 の表示に含まれるようになりました。[Fraczkiewicz, 2023; 論文投稿済].
注意:イオン化ディスクリプターが変更されたため、ADMET Predictor にある他のモデルのほとんどは、このバージョンに合わせて再トレーニングされました。そのため、予測値には若干の違いを生じることをご理解ください。
本バージョンでは、分子の形状とファーマコフォア特徴の類似性の組み合わせに基づいて 3D バーチャルスクリーニングを実行する機能が新たに搭載されています。ユーザーが 1 つ以上の参照 3D 構造を指定すると、ソフトウエアはその参照化合物とユーザーが作成した 3D コンフォーマーデータベースに含まれる化合物との間の 3D 類似性スコアを計算します。このようなデータベースの作成機能も本バージョンの新機能として提供されています。Nvidia 互換 GPU を用いたスクリーニングを実行するオプションも用意されており、それにより計算が大幅に高速化されます。
7 つの CYP アイソフォーム:1A2、2A6、2C8、2C9、2C19、2D6、3A4 の阻害定数(Ki)を予測する新たな回帰モデルが追加されました。
CYP 阻害の分類モデルも拡張されました。主要な CYP である 1A2、2C9、2C19、2D6、3A4 については、新しいデータを追加することで既存のモデルを改良し、マイナーな CYP である 2A6、2B6、2C8、2E1 については 4 つの新しいモデルが加わりました。
3D 類似性に基づいた新しい目的関数が利用可能になりました。ユーザーが 1 つ以上の参照 3D 構造を指定することにより、AIDD は各仮想化合物について、仮想化合物と各参照 3D 構造間の 3D 類似度スコアの最大値に等しい目的値を計算します。この新しい目的関数の使用は、スクリプトファイル AIDD_Sim3D_Params.txt によって制御されます。詳細な手順については、ユーザーマニュアルおよびこの AIDD_Sim3D_Params.txt ファイル内にある注釈を参照してください。
利用可能な場合は 3D ADMET モデルを要求することができるよう、実行設定が更新されました。このオプショ ンを呼び出す場合には、すべてのシード化合物は 3D 座標が定義済みである必要があります。
パラメータ PeffScalingOn が新たに追加され、選択した種がラットまたはマウスの場合、入力された permeability の値をスケーリングするかどうかを制御できるようになりました。以前のバージョンでは、このスケーリングは入力された permeability が ADMET Predictor のヒト Peff のモデル名(S+Peff)に設定された場合にのみ生じ、permeability が数値で指定された場合にはスケーリングは発生しませんでした。PeffScalingOn のデフォルト値は 1 であり、これは、入力された permeability が、選択された非ヒト種を反映するようにすでにスケーリングされている場合にのみ変更される必要があります。
ADMET Predictor 10.4 で導入された /run_cmd リクエストを使用して、いくつかのスクリプトファイルワークフローを実行できるようになりました。サポートされている操作には、代謝物予測、logD および溶解度対 pH プロファイルの生成、3D コンフォーマーの生成、化合物の変換(標準化など)、互変異性体の生成、原子プロパティを表現する画像の作成などがあります。
設定パラメーター FEATURE_UPFRONT_CHECKOUT が新たに加わり、サービス起動時にライセンスが即座にチェックアウトされるか(デフォルト)、個々のリクエストごとに必要に応じてチェックアウトされるかを制御できるようになりました。
getstatus リクエストが、ジョブ ID 無しでサブミットできるようになりました。この場合、レスポンスには、どのジョブが完了したか、キューにあるか、または現在実行中かが示されます。
predict_admet リクエストは、新しいパラメータ use3d をサポートします。これを真(true)に設定すると、利用可能な場合、予測は 3D モデルに基づいて行われます。この機能は、入力構造が 3D 座標を持つ必要があります。
以下のスクリプトファイルワークフローが追加されました:
二重結合周りの立体化学を含んでいる大環状分子の取り扱いが大幅に改善されました。
明示的な水素原子の追加と削除を行う新機能が導入されました。
複雑な環系の座標を生成する際に、ディスタンスジオメトリー試行の回数を増やすオプションが新たに追加されました。
カスタムな SMARTS パターンを持つファイルを用いて、二面角の設定を拡張することができる新しい高度なオプションが加わりました。
最も重要なバグの修正は以下の通り。
[C@@H]12C[C@H]3C[C@@H](C1)C[C@@H](C2)C3
以上
更新・新機能については、以下にある、ADMET Predictor® 10.4 (Windows版)のリリースノート: ADMET Predictor® 10.4 - May 23, 2022 でご確認ください。
以上
配座異性体を発生される機能が搭載され、ユーザーインターフェイスおよびコマンドラインのいずれからも利用することができるようになりました。単一コンフォマーおよび複数コンフォマー、MMFF力場に基づく最適化、ユーザー定義テンプレートを用いた束縛を与える座標発生などのオプションが準備されています。これにより、3次元の分子および原子ディスクリプターを用いて導かれた ADMET Predictor のプロパティ予測の利用が容易になりました。
HTPK モジュールの施された改良点は以下のとおりです:
11の細菌変異性 Ames モデル:MUT_97+1537、MUT_m97+1537、MUT_98 MUT_m98、MUT_100、MUT_m100、MUT_102+wp2、MUT_m102+wp2、MUT_1535、MUT_m1535、MUT_NIHSが、NIHS による Ames/QSAR 国際チャレンジプロジェクトから得られた新たな独自データを取り入れて再構築されました。TA98 の相対的な重要性を説明する MUTx_Risk モデルや TA98、TA100、TA1535 株およびそれらの相互作用が、新規 Ames モデルについて調整されました。MUT_Risk ウェイトは変更されていませんが、 個々の菌株での予測結果が異なるため、MUT_Risk スコアも変わる可能性があります。全体として、これらの更新は Ames モデルの化合物空間領域を大幅に拡張し、ベンチマークである NIHS、Hansen、Congying のデータセットの感度と特異性が改善されました。
通常の ADMET Predictor コマンドライン構文を用いてリクエストを送信するために新しいワークフロー (URI: /run_cmd) が追加されました。計算はサーバー上で起こり、コマンドラインから ADMET Predictor を実行して生成されされるものと同じ結果ファイルを生成しますが、実行の度にモデルを再ローディングするオーバーヘッドがありません。この新たな機能によって、ユーザー指定の pH 値における優勢なイオン化状態の予測など、これまで欠けていた機能が利用可能になりました。
一定投与および投与最適化ワークフローの両方で、血中濃度-時間プロファイルを収集できるようになりました。ネットワーク帯域幅を狭めるため時間点のサンプリングを行うオプションもあります。
以下のワークフロースクリプトファイルが追加されました。
以下の重要なバグをフィックスしました.
以上
更新・新機能については、以下にある、ADMET Predictor® 10.3 (Windows版)のリリースノート: ADMET Predictor® 10.3 - Oct 4, 2021 でご確認ください。
以上
今回のリリースでは、プログラム全体で大幅な機能強化が行われています。ここでは、変更の概要について説明しますが、すべての変更の詳細については、ADMET Predictor® ユーザーマニュアルの関連セクションを参照してください。
MedChem Studio ライセンスが、少なくとも1つの他の ADMET Predictor モジュールのライセンスを持つすべてのお客様に無償で提供されるようになりました。MedChem Studio モジュールには、化合物をスキャフォールドベースのファミリーに体系化し、R基分析を行ない、一致する分子のペアを生成する機能があります。
この変更により、これまで MedChem Studio モジュールに含まれていた化合物デザインの機能は AIDD モジュールに含まれることになり、使用するには AIDD ライセンスが必要になります。
本バージョンから、ADMET プロパティ予測、代謝産物予測、薬物動態シミュレーションなど、ADMET Predictor プラットフォームの主要な機能にアクセスするための Web API が新たに提供されます。APIは、お客様サイトにある専用 ADMET Predictor サーバー上で実行される Windows サービスアプリケーションとして実装されます。サードパーティのクライアントアプリケーションは、JSON データフォーマットの HTTP メッセージを使用してネットワーク経由でこのサービスと通信します。データフォルダー(C:¥ProgramData¥Simulations Plus, Inc¥ADMET_Predictor10.3)の下にある ADMETPredictorService サブフォルダーにある PDF 資料を参照してください。
AIDD が、各最適化サイクル中に生成される新規化合物を用いて自動的に ADMET Predictor よって呼び出されるユーザー定義の外部アプリケーションを使用して計算される目的の指定をサポートするようになりました。
ドッキングプログラム、Python や R スクリプトなどの外部アプリケーションは、ADMET Predictor データフォルダーにある特別なスクリプトファイル AIDD_ExternalApp_Params.txt 内で指定されます。詳細は、ユーザーマニュアルおよび AIDD_ExternalApp_Params.txt 内の註釈を参照してください。
また、このバージョンでは、コマンドラインから AIDD を実行するための新しいスクリプトファイルワークフローも準備されています。
このバージョンでは、CYP基質分類モデルおよび原子部位モデルへのメジャーアップデートがあります。これらの更新されたモデルは。最新の治療に関するFDA 申請レビューおよび公開文献から抽出された新しいデータに基づいています。キュレーションを広範囲に行うことによりデータが高品質なものとなり、モデル全体の性能が改良されました。
新たに2つのワークフローのためのスクリプトファイル: ms_run_AIDD.txt および ms_generate_atomprop_images.txt が実装されました。前者は、AIDD ワークフローを用いて、新規化合物を生成します。後者は ADMET Predictor の Atomic Properties ウインドウで利用可能な同じ原子プロパティのイメージファイルを生成します。詳しくは、スクリプトファイルにある注釈を参照してください。
代謝産物を予測するスクリプトファイルワークフロー ms_generate_metabolites.txt に、MedChem Designer の分子描画アプリケーションでの代謝産物表示と類似した、代謝ツリーのイメージを生成するためオプションが新たに加わりました。詳しくは、スクリプトファイルにある注釈を参照してください。
-y コマンドラインオプションを使用して書き出された回帰の不確かさ推定値が、ADMET Predictor ユーザーインターフェイスと一致して、1σ の上下界として表現されるようになりました。
Cp-time プロファイルを書き込むための -cpt コマンドラインオプションが、エクスポートされる時点数の制限に用いる任意の数値を受け入れるよう拡張されました。1 より大きい数値はエクスポートするポイント数を示し、0 と 1 の間の数値はその合計の割合を示します。
以下が修正されました。
以上
更新・新機能については、以下にある、4月28日付でリリースされた ADMET Predictor® 10.2 (Windows版)のリリースノート: ADMET Predictor® 10.2 - April 28, 2021 でご確認ください。
以上
今回のリリースでは、プログラム全体で大幅な機能強化が行われています。ここでは、変更の概要について説明しますが、すべての変更の詳細については、ADMET Predictor® ユーザーマニュアルの関連セクションを参照してください。
大手製薬会社とのコラボレーションを通じていくつかの機能強化が行われました。
以前に表示された Cp-time プロファイルに新たなプロファイルを追加表示することができるようになりました。これは、パラメーター変更の影響を視覚化するのに役立ちます。
最近の文献や Genotypic Testing に関する国際ワークショップの要旨からの調査結果に促される形で、既存の MUT_Risk を補完する変異原性リスク用の新しいモデル MUTx_Risk が加わりました。新しいモデルのリスクウェイトは、TA98、TA100、TA1535 株の相対的な重要性を反映するように厳密にキャリブレーションされており、モデルにはそれらの間での相互作用をより適切に捉えるように新しいルールが組み込まれています。これらの変更により、ベンチマークである Hansen データセットの感度と特異性の両方が向上しました。
マルチコア CPU を活用することで、いくつかの機能が大幅に高速化されました。
デフォルトでは、使用可能なすべてのコアから 1 つを除いたコアを計算に使用しますが、FILE >> Preferences >> Advanced タブで変更することができます。
いくつかのコマンドライン機能が新たに追加されました。これらはすべて、スクリプトファイルによるワークフローの仕組みを用いるものであり、ADMET Predictor® は、実行制御パラメーターを含む、変更可能なテキストファイルを渡すことによって呼び出されます。新たに加わったワークフローを以下に示します。それぞれの詳細については、関連するスクリプトファイルとユーザーマニュアルに記載されています。
これらやその他のスクリプトファイルワークフローは、コマンドラインからの引数の指定をサポートするようになりました。例えば、ms_generate_metabolites.txt の structureFile パラメータは、次のようにコマンドラインで指定することができます。
ADMET_Predictor.exe ms_generate_metabolites.txt -structureFile MyFile.sdf
これにより、ファイルで定義されている他のパラメーターを残したまま、各実行で変更される傾向のあるパラメーターを、コマンドラインで指定することができます。
以下のような不具合が修正されました:
以上
更新・新機能については、以下にある、9月25日付でリリースされた ADMET Predictor® X (Windows版)のリリースノート: ADMET Predictor® X - September 25, 2020 でご確認ください。
以上
今回のリリースでは、プログラム全体で大幅な機能強化が行われています。ここでは、変更の概要について説明しますが、すべての変更の詳細については、ADMET Predictor ユーザーマニュアルの関連セクションを参照してください。
AIDD モジュールを使用すると、ユーザーは一つ以上のシード化合物から開始する反復手順により、複数のターゲットプロパティに対して最適化された候補化合物を生成することができます。化合物は変換ルールを介して変更され、複数のターゲットプロパティに対して評価されます。その結果、1つ以上のターゲットプロパティに対してパレート最適である化合物のライブラリが作成されます。これにより、ユーザーは、各ライブラリの任意の重みを選択せずに最終ライブラリでターゲットプロパティに対して相互にトレードオフできます。合成難易度や ADMET リスクなどのターゲットプロパティを組み込み、範囲外のペナルティを使用することで、ほかの de novo 設計アルゴリズムでよく見られる、問題のある化合物の頻度を減らすことができます。さらに、スキャホールドと任意のフィルター規則を定義する機能は、アルゴリズムがケミカルスペースによる望ましくない領域探索を行うことの防止に役立ちます。ターゲットプロパティとして HTPK プロパティ (%Fa または %Fb) を含めると、結果として得られる化合物ライブラリにおいて、良好な薬物動態特性をターゲットに設定することができます。したがって、この新しいモジュールは、ADMET Predictor の多くの機能を柔軟かつ合理化されたプロトコルに組み合わせて、複数のターゲットプロパティに対して同時に最適化された仮想化合物を迅速に生成します。
臨床的に重要であるトランスポーターに焦点を当てた新しいモジュールです。全 24 種類のモデル (18 種類の新しいモデル) を搭載しています。世界各国の規制当局が発行しているガイダンスで明示されている、9 種類の重要なトランスポーターの基質、阻害剤、Km 予測のほぼ完全なマトリックスをカバーしています:P糖タンパク質 (P-gp)、乳がん耐性タンパク質 (BCRP)、有機アニオン輸送ポリペプチド (OATP1B1, OATP1B3)、有機アニオン (OAT1, OAT3)、有機カチオン (OCT1, OCT2)、胆汁酸塩輸出ポンプ (BSE)。
ADMET Predictor のマルチコア CPU を活用する機能が拡張され、ADMET プロパティ予測と HTPK シミュレーションで使用できるようになりました。以前のバージョンと同様、グラフィカルユーザーインターフェイスの起動時に ADMET Predictor 10.0 は、使用可能なスレッドの総数を決定します。ほとんどのコンピューターにおいて、スレッドは物理コアの数の 2 倍です (4 コアマシンの場合は 8 スレッド)。デフォルトでは使用可能なスレッドの 1 つを除いてすべて計算に使用しますが、この設定は変更可能です。コマンドラインから実行する場合、スレッド数は新しい -N フラグを使用して明示的に設定する必要があります。このフラグを省略すると、単一スレッドのみが計算に使用されることになります。
マルチスレッドを使用して得られる速度向上は、コンピューターのハードウェアと分析される特定のデータセットに依存しています。Simulations Plus 社のテストでは、8 以上のスレッドを使用した場合、実行時間を少なくとも 4 倍または 5 倍短縮できることが示されました。
マルチスレッドをサポートする追加機能には、合成難易度スコアの計算やクエリを使用した外部ファイルのスクリーニングがあります。
大手製薬会社とのコラボレーショにより、HTPK モジュールにいくつかの機能強化が行われました。
ヒトとラットの肝細胞クリアランスを予測するための 2 つのモデル (HEP_hCLint, HEP_rCLint) と、リコンビナント CYP クリアランス の合計を提供するモデル (CYPSum_CLint) が新たに搭載されました。
データの追加により以下のモデルが改善されました。
リスクモデルに "Out-of-scope Factor" を指定する新しいオプションが追加されました。リスクルールが特定の化合物に対して範囲外のモデルを使用し、そのモデルの結果がルールの評価に影響を与える場合、そのルールの重みは "Out-of-scope Factor" で乗算されます。デフォルトでは 0.5 に設定されており、File >> Preferences >> ADMET から変更することができます。通常、ルールの評価によりリスクスコアが増加されますが、範囲外のモデルが結果に影響を与える場合、"+" がルールコードに追加されます。同様に、ルールが範囲外のモデルによってリスクスコアの増加を引き起こすわけではないが、そのルール評価が範囲外のモデルの影響を受ける場合、"-" がルールコードに付加されて出力されます。
ADMET_Risk、関連するリスクモデルは、これらの変更や WDI データセットへの追加キュレーション、様々な ADMET モデル の更新を考慮して再評価され、その結果、ルールで使用されるカットオフの多くが変更されました。
MedChem Designer には、化学構造をスキャフォールドクエリに変換して、AIDD や Combinatorial Transform などの ADMET Predictor の構造デザインで使用できる新しい機能が追加されています。これは、デザイン変更が化合物構造の指定された領域に対してのみ行われるようにするために特に役立ちます。
Combinatorial Transform 機能が強化されました。新しい変換ルールがデフォルトファイル “moleculeTransforms.crf” に追加され、既存のルールはより構造的に特化されています。スキャフォールドクエリを指定するプロセスが簡略化されました。望ましくない類似体をさらに除去するのを助けるために “DrugLikeFilters.cqf” という名前のファイルが提供されています。非生産的な変換経路を終了する新しいオプションは、著しい速度改善をもたらします。
スプレッドシートのヒートマップのカラーリング、Classes タブ内のカラムヒストグラム、R Group Analyzer のスプレッドシートのカラーリングでログスケーリングがサポートされるようになりました。カラースケールを変更するには、Compounds タブのカラムヘッダーを右クリックし、コンテキストメニューから Set Color Scale >> Log を選択します。カラムヒストグラムのログスケーリングは、これらのカラムを追加、変更するときに表示されるオプションダイアログを使用して指定できます。
View >> Hide/Unhide >> Clear Control Panel Filters
View >> Hide/Unhide >> Reset Control Panel Filter Ranges
どちらのオプションも範囲をリセットして Unhide を実行しますが、2番目のオプションはコントロールパネルで現在選択されている属性を保持します。
重要な Bug fixes は以下の通りです。
以上
今回のリリースでは、MedChem Studio モジュールやADMET Modeler モジュールのみならず HTPK Simulation モジュールへの大幅な機能強化が加えられています。主要なクリアランスメカニズムを予測するためにいくつかの新しいモデルが追加されています。さらに、ユーザーインターフェイスの改良と多くのバグフィックスが行われています。
ADMET Predictor 9.5 がFlexeraライセンスサーバーと通信するためには、Simulations Plus ベンダーデーモン(simplus.exe)の最新バージョンをライセンスサーバーにインストールする必要があります。この新しいデーモンは、ADMET Predictor 9.5 インストールパッケージに付属しています(インストーラー内の Vendor Daemon フォルダー)。以下の「ベンダーデーモンの置き換えについて」を参照してください。
初めてADMET Predictor 9.5を使用する前にこれらの作業を行うよう、ライセンス管理者に相談してください。
新たに加わったStructure Sensitivity Analysis(構造感度分析:SSA)ウィンドウは、ある予測プロパティへの原子の寄与をインタラクティブに視覚化します。例えば、リード化合物の、どのフラグメントが予測毒性に最も寄与するのかを調べることが可能になります。この機能は、既存の Descriptor Sensitivity Analysis(DSA)ウィンドウを補完するものであり、化学的に直観で理解できる方法で局所的な原子の感度情報を提供します。現在、21のモデルをこのウィンドウで表示することが可能であり、ユーザー自身のモデルを構築することもできます。SSAウィンドウは、ADMET Predictor、MedChem Designerのどちらからでも呼び出すことが可能です。
多くの回帰モデル予測に、各予測の予測誤差期待標準偏差を示す不確実性評価を伴うようになりました。これらの評価は、組み込みのADMET PredictorモデルおよびADMET Modelerを用いて構築されたユーザーモデルのどちらについても提供され、すでにクラス分類モデルで提供されている信頼性評価を補完するものです。
回帰モデルのパフォーマンスプロットに、予測の不確実性評価に対応するエラーバーを表示するオプションが搭載されました。Model Editorウィンドウでは、新たに、エクスポートとインポートのオプションが提供され、ユーザー組織内でのユーザーモデルの共有を容易にします。Model Performance Gridには、新たな計量、RMSUが加えられ、回帰モデルの根平均二乗不確実性を表示します。ModelerおよびPerformance Plotウィンドウは、より小さなサイズに変更することが可能になりました。
回帰不確実性モデルの解析を容易にする、新たなプロットタイプが加わりました。これは、分類信頼モデルのConfidence Analysis プロットに匹敵するものです。これらには、Squared ErrorとEnsemble Standard Deviationの累積分布プロット、同量Quantile - Quantile(Q-Q)プロット、Normalized ErrorのQ-Qプロット(観測モデル誤差を不確実性推定値で割ったもの)が含まれます。
その他のパフォーマンスプロットへの変更点として以下があります:
MedChem DesignerにADMET Predictorからのいくつかの予測と表示の機能が加わりました。これらには、pKa Microstates ウィンドウやAtomic Properties ウィンドウ、logD と溶解度対pH曲線、%F/%Fb予測やCp-time 曲線などのHPTK機能などがあります。
その他に重要な変更点として:
薬理学的に関連のあるトランスポーターについて新たに3つのモデル:乳癌耐性タンパク質(BCRP; substrate/nonsubstrate)、 有機カチオントランスポーター 2 (OCT2; inhibition) 、胆汁酸塩輸送ポンプ(BSEP; inhibition)が加わりました。また、日本のNIHS(国立医薬品食品衛生研究所)からのデータを用いて構築された、新しいAmes変異原性モデルがあります。また、アルデヒド酸化酵素(AOX)基質分類モデル、AOX代謝の可能性がある原子サイトを予測するモデルがあります。
新しいデータの追加により、以下のモデルが改良されました。
ADMET Predictor および MedChem Designer に、CYP酵素に加え、AOX、UGT、エステラーゼの酵素が代謝産物予測に加わりました。予測では、利用可能であれば、既存の基質モデルとサイトモデルを利用します。例えば、UGT代謝物予測では、既存のアイソファーム特異的なUGT基質モデルを、また、AOX代謝産物予測では、新規AOX基質モデルとサイトモデルを取り入れます。また、予測では、関連する代謝変換の文献から得られた例に由来するエキスパートルールも利用されています。これは、予測される代謝産物が、観測される可能性が最も高い代謝産物に限定されることを確実にする手助けになります。
以下の重要なバグに対応しました:
以上
今回のリリースでは、MedChem Studio モジュールやADMET Modeler モジュールのみならず HTPK Simulation モジュールへの大幅な機能強化が加えられています。主要なクリアランスメカニズムを予測するためにいくつかの新しいモデルが追加されています。さらに、ユーザーインターフェイスの改良と多くのバグフィックスが行われています。
ADMET Predictor 9.5 がFlexeraライセンスサーバーと通信するためには、Simulations Plus ベンダーデーモン(simplus.exe)の最新バージョンをライセンスサーバーにインストールする必要があります。この新しいデーモンは、ADMET Predictor 9.5 インストールパッケージに付属しています(インストーラー内の Vendor Daemon フォルダー)。以下の「ベンダーデーモンの置き換えについて」を参照してください。
初めてADMET Predictor 9.5を使用する前にこれらの作業を行うよう、ライセンス管理者に相談してください。
新たに加わったStructure Sensitivity Analysis(構造感度分析:SSA)ウィンドウは、ある予測プロパティへの原子の寄与をインタラクティブに視覚化します。例えば、リード化合物の、どのフラグメントが予測毒性に最も寄与するのかを調べることが可能になります。この機能は、既存の Descriptor Sensitivity Analysis(DSA)ウィンドウを補完するものであり、化学的に直観で理解できる方法で局所的な原子の感度情報を提供します。現在、21のモデルをこのウィンドウで表示することが可能であり、ユーザー自身のモデルを構築することもできます。SSAウィンドウは、ADMET Predictor、MedChem Designerのどちらからでも呼び出すことが可能です。
多くの回帰モデル予測に、各予測の予測誤差期待標準偏差を示す不確実性評価を伴うようになりました。これらの評価は、組み込みのADMET PredictorモデルおよびADMET Modelerを用いて構築されたユーザーモデルのどちらについても提供され、すでにクラス分類モデルで提供されている信頼性評価を補完するものです。
回帰モデルのパフォーマンスプロットに、予測の不確実性評価に対応するエラーバーを表示するオプションが搭載されました。Model Editorウィンドウでは、新たに、エクスポートとインポートのオプションが提供され、ユーザー組織内でのユーザーモデルの共有を容易にします。Model Performance Gridには、新たな計量、RMSUが加えられ、回帰モデルの根平均二乗不確実性を表示します。ModelerおよびPerformance Plotウィンドウは、より小さなサイズに変更することが可能になりました。
回帰不確実性モデルの解析を容易にする、新たなプロットタイプが加わりました。これは、分類信頼モデルのConfidence Analysis プロットに匹敵するものです。これらには、Squared ErrorとEnsemble Standard Deviationの累積分布プロット、同量Quantile - Quantile(Q-Q)プロット、Normalized ErrorのQ-Qプロット(観測モデル誤差を不確実性推定値で割ったもの)が含まれます。
その他のパフォーマンスプロットへの変更点として以下があります:
MedChem DesignerにADMET Predictorからのいくつかの予測と表示の機能が加わりました。これらには、pKa Microstates ウィンドウやAtomic Properties ウィンドウ、logD と溶解度対pH曲線、%F/%Fb予測やCp-time 曲線などのHPTK機能などがあります。
その他に重要な変更点として:
薬理学的に関連のあるトランスポーターについて新たに3つのモデル:乳癌耐性タンパク質(BCRP; substrate/nonsubstrate)、 有機カチオントランスポーター 2 (OCT2; inhibition) 、胆汁酸塩輸送ポンプ(BSEP; inhibition)が加わりました。また、日本のNIHS(国立医薬品食品衛生研究所)からのデータを用いて構築された、新しいAmes変異原性モデルがあります。また、アルデヒド酸化酵素(AOX)基質分類モデル、AOX代謝の可能性がある原子サイトを予測するモデルがあります。
新しいデータの追加により、以下のモデルが改良されました。
ADMET Predictor および MedChem Designer に、CYP酵素に加え、AOX、UGT、エステラーゼの酵素が代謝産物予測に加わりました。予測では、利用可能であれば、既存の基質モデルとサイトモデルを利用します。例えば、UGT代謝物予測では、既存のアイソファーム特異的なUGT基質モデルを、また、AOX代謝産物予測では、新規AOX基質モデルとサイトモデルを取り入れます。また、予測では、関連する代謝変換の文献から得られた例に由来するエキスパートルールも利用されています。これは、予測される代謝産物が、観測される可能性が最も高い代謝産物に限定されることを確実にする手助けになります。
以下の重要なバグに対応しました:
以上
今回のリリースでは、MedChem Studio モジュールやADMET Modeler モジュールのみならず HTPK Simulation モジュールへの大幅な機能強化が加えられています。
主要なクリアランスメカニズムを予測するためにいくつかの新しいモデルが追加されています。さらに、ユーザーインターフェイスの改良と多くのバグフィックスが行われています。
ADMET Predictor 8.5 で導入されたHTPK(high-throughput pharmacokinetic)Simulation モジュールは、肝臓と腎臓のクリアランスで強化された単一中央コンパートメントに関連する、GastroPlus™ で使用されている基本ACAT™ 消化管吸収シミュレーションの手法を用いて消化管吸収率(%Fa)及び相対的バイオアベイラビリティ(%Fb)を推定します。バージョン9.0では、薬物動態パラメーター Cmax、Tmax、AUC の推量が追加されました。これらは、%Fa や %Fb の計算時に出力されます。
化合物の血漿中濃度を時間の関数として表示する機能が新たに追加されました。これらの濃度曲線は、GastroPlusによるコンパートメントシミュレーションから得られる曲線と定量的に非常によく一致しています。
化合物の溶解度と透過性の変化に対する %Fa、%Fb、あるいは他のPKパラメーターの感度を評価するための新機能も追加されました。GastroPlus™ に搭載されている同様のパラメーター感度分析(PSA)機能と同じく、これにより、最適な暴露量に達するためにこれら2つの物理化学的特性のどちらを改善する必要があるかを評価することができます。
MedChem Studio™ モジュールには、キーの生成と視覚化のための新たな機能が追加され、これらのキーは、利用可能な手法の1つとしてよく用いられている Extended-Connectivity Fingerprints(ECFP)を含むサブストラクチャベースのディスクリプターの特別なクラスとなります。
これら及び他のタイプのキーは、ADMET Predictor 内での様々な用途を持ち、SARの発見やプロパティに関する構造アラート(例えば、アッセイ干渉、反応性代謝物、急性毒性)の視覚化などに使われます。ユーザーは化合物プロパティと最も関連性の高いキーを簡単に識別することができるので、SARをより視覚的に解釈可能にするのに役立ちます。
logDと溶解度を pHの関数として表示するためのグラフ表示が新たに追加されました。この機能は、同社のソフトウェア GastroPlus™ では以前から利用可能であったものです。
他の大きな変更点として以下があります:
独自の分子荷電モデルと、Varmaらのデータをベースにした新しいMDCK-LE(MDCK-limited efflux)透過性分類モデルを使用した、拡張クリアランス分類システム(Extended Clearance Classification System:ECCS)が実装されました。代謝、腎臓排泄、肝臓摂取によるクリアランスについて3つの二項分類モデルとともに、これら3つの経路のうちのどれが最も支配的なクリアランスである可能性が高いかを予測するための3項ANNEモデルが追加されました。マニュアルのADMET Properties の章に、Pharmacokinetic Properties に関する新しいセクションが追加され、異なるクリアランスモデル間の相互作用について説明しています。
生物薬剤学分類システム(BCS)および開発可能性分類システム(DCS)に準じて、化合物がどのように分類されるかを示すグラフィック表示が可能になりました。このインターフェースは非常に柔軟な設定が可能であり、化合物の開発を成功に導くために化合物の投与量、溶解度、透過性がどのように役割を果たすかを対話的に調べる便利なツールです。
ニューラルネットワークアンサンブル(ANNE)を使用してマルチクラス(3つ以上)分類モデルの構築が可能になりました。これまでは、サポートベクターマシン(SVM)モデルを使用してのみ可能だったものです。
Modeler の Variable Selection ページにあるオプションの ”Reference Variable” を導入することにより、DELTA Models を構築するためのワークフローの大幅な簡素化と効率化が図られました。詳しくはマニュアルを参照してください。
他の重要な変更点として以下があります:
重要なバグフィクスについて以下に記載しています。
以上
今回のリリースでは、MedChem Studio モジュールやADMET Modeler モジュールのみならず HTPK Simulation モジュールへの大幅な機能強化が加えられています。
主要なクリアランスメカニズムを予測するためにいくつかの新しいモデルが追加されています。さらに、ユーザーインターフェイスの改良と多くのバグフィックスが行われています。
ADMET Predictor 8.5 で導入されたHTPK(high-throughput pharmacokinetic)Simulation モジュールは、肝臓と腎臓のクリアランスで強化された単一中央コンパートメントに関連する、GastroPlus™ で使用されている基本ACAT™ 消化管吸収シミュレーションの手法を用いて消化管吸収率(%Fa)及び相対的バイオアベイラビリティ(%Fb)を推定します。バージョン9.0では、薬物動態パラメーター Cmax、Tmax、AUC の推量が追加されました。これらは、%Fa や %Fb の計算時に出力されます。
化合物の血漿中濃度を時間の関数として表示する機能が新たに追加されました。これらの濃度曲線は、GastroPlusによるコンパートメントシミュレーションから得られる曲線と定量的に非常によく一致しています。
化合物の溶解度と透過性の変化に対する %Fa、%Fb、あるいは他のPKパラメーターの感度を評価するための新機能も追加されました。GastroPlus™ に搭載されている同様のパラメーター感度分析(PSA)機能と同じく、これにより、最適な暴露量に達するためにこれら2つの物理化学的特性のどちらを改善する必要があるかを評価することができます。
MedChem Studio™ モジュールには、キーの生成と視覚化のための新たな機能が追加
され、これらのキーは、利用可能な手法の1つとしてよく用いられている Extended-Connectivity Fingerprints(ECFP)を含むサブストラクチャベースのディスクリプターの特別
なクラスとなります。
これら及び他のタイプのキーは、ADMET Predictor 内での様々な用途を持ち、SARの発見やプロパティに関する構造アラート(例えば、アッセイ干渉、反応性代謝物、急性毒性)の視覚化などに使われます。ユーザーは化合物プロパティと最も関連性の高いキーを簡単に識別することができるので、SARをより視覚的に解釈可能にするのに役立ちます。
logDと溶解度を pHの関数として表示するためのグラフ表示が新たに追加されました。この機能は、同社のソフトウェア GastroPlus™ では以前から利用可能であったものです。
他の大きな変更点として以下があります:
独自の分子荷電モデルと、Varmaらのデータをベースにした新しいMDCK-LE(MDCK-limited efflux)透過性分類モデルを使用した、拡張クリアランス分類システム(Extended Clearance Classification System:ECCS)が実装されました。代謝、腎臓排泄、肝臓摂取によるクリアランスについて3つの二項分類モデルとともに、これら3つの経路のうちのどれが最も支配的なクリアランスである可能性が高いかを予測するための3項ANNEモデルが追加されました。マニュアルのADMET Properties の章に、Pharmacokinetic Properties に関する新しいセクションが追加され、異なるクリアランスモデル間の相互作用について説明しています。
生物薬剤学分類システム(BCS)および開発可能性分類システム(DCS)に準じて、化合物がどのように分類されるかを示すグラフィック表示が可能になりました。このインターフェースは非常に柔軟な設定が可能であり、化合物の開発を成功に導くために化合物の投与量、溶解度、透過性がどのように役割を果たすかを対話的に調べる便利なツールです。
ニューラルネットワークアンサンブル(ANNE)を使用してマルチクラス(3つ以上)分類モデルの構築が可能になりました。これまでは、サポートベクターマシン(SVM)モデルを使用してのみ可能だったものです。
Modeler の Variable Selection ページにあるオプションの ”Reference Variable” を導入することにより、DELTA Models を構築するためのワークフローの大幅な簡素化と効率化が図られました。詳しくはマニュアルを参照してください。
他の重要な変更点として以下があります:
重要なバグフィクスについて以下に記載しています。
以上
今回のリリースでは、ADMET Predictor全体で多くの主要な機能拡張が行われています。最大の特徴は、吸収率、バイオアベイラビリティ、および目的とする血漿中濃度を達成するのに必要な投与量を推定するための次世代HTPK Simulationモジュールを搭載した点にあります。また、多数のバグ修正と共に、インターフェイス、ADMETプロパティーモデル、MedChem Studio™モジュール、MedChem Designer™、ADMET Modeler™も大幅に改良されました。
HTPK(high-throughput pharmacokinetic)Simulationモジュールでは、GastroPlus™で使用されている基本的なACAT™腸管吸収シミュレーションスキームを用いて、吸収率(Fa)および相対バイオアベイラビリティ(Fb)を推量することが可能です。これには、傍細胞透過性、胆汁塩の影響、析出現象が考慮されています。また、Fbは吸収率に推定肝クリアランスを適用して計算されます。
所定の定常血漿中濃度(OptDose)に到達するために必要となる投与量の推定には、定常状態でのヒト分布容積のANNEモデルであるVdを利用することも可能ですが、今回、GastroPlus™で使用されているメカニスティックな推定値も利用可能になりました。これらの推定値はシミュレーション実行後にスプレッドシートに表示されます(ヒトの値(S+hVd_PBPK)またはラットの値(S+rVd_PBPK)のいずれか)。
ラット血漿における非結合型薬物の割合を予測するためのモデル(rat_fup%)が、ヒト血漿に対するモデルを補完するために加えられました。ラット肝ミクロソームにおけるCYP固有クリアランスのモデル(CYP_RLM_CLint)と同様に、ラットにおける血液-血漿比のモデル(RBP_rat)も利用可能です。これらに関連するヒトおよびラットモデルの利用には、HTPK Simulationモジュールのライセンスが必要になります。
今回、ADMET PredictorにMedChem Studio™からさらに多くのインターフェイス機能が組み込まれました。例えば、メインの化合物スプレッドシートは、デフォルトである「テーブル表示」とスプレッドシートの各セルに分子構造画像が表示される「タイル表示」を切り替えることができるようになりました。また、新たに追加されたコントロールパネルにはダイナミックフィルターバーがあり、目的とする物性値の範囲から外れている化合物を非表示にすることができます。
Excelファイルへのエクスポート機能が大幅に改善されました。最も重要な点は、Excelファイルの保存に要する時間が大幅に短縮されたことです。多くの場合、100倍以上も短縮されるようになっています。また、以前の *.xlsフォーマットに加えて、最近のXMLベースの *.xlsxファイルフォーマットがサポートされるようになりました。最近のフォーマットの方は、カラムの数が256を超えてもエクスポートすることが可能です。Excelに埋め込まれた画像もスプレッドシート内の他のカラムデータとともに適切にソートされるようになり、また、「out of scope(範囲外)」となった予測値は、ADMET Predictorのインターフェイスと同じく赤いフォントで表示されるようになりました。
新たに追加された層化抽出法の機能を使用して、スプレッドシート内の化合物のサブセットを選択することができるようになりました。テストセットを選択するためのADMET Modelerの機能と同様に、指定した属性を用いてデータがソートされ、可能な限り均等に分割されるので、指定したサイズのサブセットを選択することができます。このサブセットは、ADMET Modelerの外部テストセットとして、またはADMET Predictor内の他の目的のために使用することができます。実装の詳細については、ADMET Modelerのマニュアルを参照ください。
その他の改良点は以下になります:
今回もADMET Predictor 8.5のMedChemモジュールに、MedChem Studio™のより多くの機能が追加されました。Classes >> Subclasses メニューの下に追加されたいくつかの新しいオプションを含め、サブクラスに対するサポートが強化されています。新たに追加された Classes >> Compare Classes メニューには、「クラスvsクラス」に関する様々な類似スコアを計算する機能があります。化合物に対する属性値と同様に、クラスにも任意の属性値を定義して注釈付けすることができるようになりました。ADMET Predictor 8.5では、MedChem Studio™のコマンドラインやPipeline Pilot機能もサポートされるようになりました。
複数の化合物について、その化学合成の難易度を評価する新しい機能が追加されました。ErtlとSchuffenhauerによる研究(マニュアルを参照ください)に基づき、化合物に0(容易)™10(困難)のスコアが割り当てられます。この論文は、これらのスコアが、経験豊富な合成化学者による独自の難易度推定値とよく一致していると報告しています。
R Tableタブに Find Subsets Using R Group という新しい機能が追加されました。この機能を用いて、R位置のいくつかが特定の置換基を持っている興味深い化合物群を見つけることができます。例えば、Rテーブルに5つのR基がある場合、これらの置換基のうち3つが固定(例えば、R1 = CH3、R4 = H、R5 = Cl)で、2つの置換基だけが可変であるとします。この化合物群だけで生成した新しいRテーブルは、R基が5つから2つに減るので、その後の解析が大幅に容易になります。
R Group Analyzerにも、タイル型とサークル型プロットの切り替え機能、スプレッドシートのセルに物性値をテキスト表示するオプション、フィルター用スライダーのカラー表示など、多くの改良点があります。
化学構造をクリップボードにコピーしたときに、予測されたADMETプロパティを3つまで渡すことができるようになりました。これらは、分子構造画像とともにMicrosoft PowerPointなどのアプリケーションに渡すことができます。3つのADMETプロパティは、「Change Settings」ダイアログの「Format」タブを使用して選択します。
予測されたADMETプロパティを表示するスプレッドシートは、ユーザーが指定した一部のプロパティだけに限定することができるようになりました。指定したプロパティのリストはファイルに保存されるため、Designerを使用するたびにこれらのプロパティだけが表示されます。
ADMET Predictorのスプレッドシートと同様に、Designerもヘテロ原子をタイプごとに色付けする新しいオプションが追加されました。
モデル構築を並列化するためにマルチスレッド化が実装され、アンサンブルモデルに寄与するサブモデルはそれぞれ独自のスレッドで構築されるようになりました。特にADMET Predictorからのデータを扱う場合、Modelerによるメモリ使用量が大幅に削減されました。
Confidence Analysisは、単一(統合型)ではなく、正と負の予測に分けた(分割型)ベータ二項分布へフィッティングさせることによって改善されました。ばらつきのあるデータセットに対する不確かさの分析結果では、分割型の方が統合型でみられるものよりも信頼度が高い傾向があります。プログラムでは、統合型のベータ二項分布と分割型のベータ二項分布へのフィッティングの両方を試し、各モデル構築に対して最適な方が選択されます。また、この新手法を補完する尺度としてMin ConfidenceがModel Performance Gridに追加されたことにより、モデル構築全体でのCFAの品質を一目で評価することができるようになっています。詳細はマニュアルを参照ください。
ADMET Modelerには、他にも多くの重要な変更点があります:
重大なバグ修正のいくつかを以下に説明します。
以上
今回のリリースでは、ADMET Predictor™ 全体を通して機能拡張が数多く行われています。最も大きな変更点は、バージョン8.1では32bit版と64bit版の両方が利用できるようになったことです。64bit版を利用することで、大量データの読み込みや計算処理の高速化が期待できます。(64bit版を利用するには、ライセンスデーモンも64bit版にする必要があります)。また、スプレッドシートの処理がより効率的になり、より大きなデータセットを収納できるようになりました。一方、ADMET Modeler™ はファイルをより速く読み込めるようになり、また、使いやすくなりました。
インターフェイス、ADMET物性モデル、MedChem Studio™、ADMET Modeler™ に関するその他の改良点は以下で詳しく説明します。
Add compound attributes の Select from list... オプションに Sum of CYP Clearances と ADMET Risk Codes が追加されました。前者は個々のCYP CLintの予測値の総和を計算し、後者はADMET Risk 定義ファイルにある各ルールに対するバイナリー属性を生成します。
ADMET Predictor™ 8.0 では、ヒートマップのカラースケールを増加する方向から減少する方向に反転させることで、対応するスタープロットのくさびも切り替わりましたが、8.1 ではヒートマップの設定がスタープロットに影響しないように変更されました。これにより、大きな値があまり望ましくない場合がある ADMET Risk™ のような属性表示の解釈が容易になりました。
Shiftキーを押しながらヒストグラムのバーをクリックすることで、それに相当するスプレッドシートの行が選択されるようになりました。Ctrlキーを押しながらバーをクリックしていくと複数の行を選択することができます。また、ヒストグラムプロットの属性として対応する Risk Code を選択することにより、ADMET Risk™ ルール違反の分布を視覚化できるようになりました。
信頼性予測を提供する in silico の Ames 試験モデルが新たに追加され、以前のモデルよりも広範囲に適用可能になりました。また、CYP3A4_Substr モデルは、信頼性評価を含んだ、広い適用領域をもつモデルに置き換えられました。
ADMET Predictor™ 8.0では、肝毒性に対して二つのADMET Risk™ルール(Hp, SG)がありました。市販薬に関する予測試験に基づき、これらの2つのルールが1つのルール(HEPX)に統合されました。このルールでは、WDIの2270化合物のうち15.5%がリスクありと予測されたのに対し、旧ルールではさらに多くの化合物がリスクありと予測されていました。
どの細菌株が変異原性を有すると予測されるかをより明確にするため、MUT Risk ルールが単純化されました。特に、これまでのS2とm2ルールは4つのルール(S_98, m_98, S100, m100)に分離されました(それぞれの重みは0.5)。
グルタチオン、コエンザイムA、システイン、ホモシステイン、システアミンなどに含まれるCHCH2SHのようなメルカプトエチル基を、CYPによる酸化の可能性のある部位として考慮しないようになりました。そのような酸化で生じるスルフィン酸生成物を逆に還元する in vivo の系は、肝細胞中のNADPHやグルタチオンを急速に排除し、致命的な結果になるでしょう。
このことからあらゆる哺乳類CYPがそのような化合物を攻撃することに対する非常に強い進化的選択圧があることがわかります。
薬物設計で使用するコンビナトリアルな置換基操作や骨格置換操作が追加されました。同等な機能はMedChem Studio™自体にはありましたが、ADMET Predictor 8.0のMedChem Studioモジュールにはありませんでした。
RTables メニューに Add Group Attributes オプションが追加されました。このオプションを選択するとSMILES文字列を値として持つ化合物属性 R1_Label や R2_Label などが追加されます。
R Group Analyzer でセッションをディスクに保存するときやエクセルにエクスポートするときに、Rn x Rm ページと Rn x Rn ページの行と列の順序が保持されるようになりました。さらに、行と列は分子量でソートしたり、ドラッグ&ドロップで並び替えることができるようになりました。
R Table Explosion を使用したとき、元のスプレッドシートにある化合物と一致する生成物には、それらの生成物を除外するオプションがオフになっていても簡単に識別できるようにラベルが付くようになりました。
コメントを追加することにより、クラスに注釈をつけることができるようになりました。
データセットの再読み込みがより高速化されました。また、プロジェクトに1000以上のデータポイントがある場合、Modeler ウィンドウの下部にあるステータスバーに読み込みの進行状況が表示されるようになりました。
Kohonenマッピングを使用するプロジェクトを開くと、Test Set Selection が Reuse an existing Kohonen map にセットされるようになりました。Kohonenマップの生成自体は約2倍ほど高速化されましたが、中規模から大規模なデータセットに対しては、既存のマップを再利用することが適切な判断であり、いまだにデフォルトのオプションとなっています。また、SOMファイルにあるセルの平均値出力が正しく動作するようになりました。
乱数シードの扱いがより強固なものになりました。特に、モデル構築中にPNO表示ウィンドウを起動しても、その後のモデル構築には影響しなくなりました。さらに、すべてのアーキテクチャが「見ている」乱数シードは、モデルの実行とアーキテクチャーに提供された最初のシードによってのみ決定されるようになり、モデルが埋め込まれているグリッドサイズはもはやプロセスに影響を与えなくなりました。ただし、遺伝的アルゴリズム(GA)により選択されたディスクリプターを用いて構築されるモデルはこのルールの例外になるので、その場合の結果は、依然としてグリッド設定に依存します。このため、バージョン8.0以前で得られたモデル構築の結果が、バージョン8.1を用いても正確には再現されない場合があり、異なる乱数シードを使用したかのような挙動を示すことになります。
バージョン8.0では、検証プールがモデル構築へ関与する前に早期終了になった場合にも、”verify”の統計値がPerformance Gridに表示されていました。そのような場合(例えば、平均化や閾値の調整を用いたANNE分類モデル)、”train”および”verify”の統計値は同一でした。このバージョン8.1では、冗長な”verify”エントリーは省略され、”train”の統計値はそのトレーニング set だけからでなく、トレーニング pool 全体から計算されます。
ユーザーは、モデル構築プロセスの最初のステップとしてプロジェクト名を入力するよう求められるようになりました。デフォルト名は従属変数の名前、日付、および現在のセッションで作成されたモデルの数に基づいて付けられますが、ユーザーが自由に独自のものを付けることができます(例えば、ノートの参照番号に連番をふったものなど)。それらは Modeler での作業をディスクに保存するときに、その選択を確認するように求められます。
Every n-th テストセット選択オプションが、Stratified sampling と呼ばれる、より柔軟な別の方法に置き換えられました。観測値は指定した値(分類モデルの場合は、通常、従属変数)でソートされ、分位数に分割されます。各分位数のある観測値は削除され、テストセットに入ります。ユーザーは、削除する観測値をランダムに選択するのか、分位数の中央値にするのかを指定することができます。Every n-th では、各分位数の最初の観測値がテストセットに入っていました。詳細はマニュアルを参照ください。
以前はANNだけだった Autofill ボタンが、各モデル構築手法の Settings ページで利用できるようになりました。
一般的な先頭文字の組み合わせ(”No”、”NEG”、”LOW”、”inactive”)が認識されるようになり、デフォルトでネガティブの分類として選択されるようになりました。大文字と小文字の区別はありません。
バージョン8.0では、ニューロンの最大数を指定してモデルが構築され、そのユーザーが指定した制限を超えない範囲で調整可能な重みの数が決まっていました。例えば、ステップ数が2でニューロン数が3〜6の場合、3、5、6個のニューロンのモデルになりました。そのようなプロジェクトは保存することはできましたが、再度読み込むことができませんでした。今回、この問題が修正されました。
平均化を用いて構築されたANNE分類モデルに対する閾値調整により、ポジティブとネガティブの分類が交換されたときに対称性をもつ結果となるようになりました。これは、8.0以前のバージョンには当てはまりません。
Modeler は、Performance Grid の左上にあるXを使ってウィンドウを閉じたときにも、STOPボタンを押したときと同様に、正しく停止するようになりました。
分類モデルの構築時には Logify オプションの設定が自動的に無視されるようになりました。
以上
ADMET Predictor™ 8.0 では、デフォルトのインストール場所やファイルの保存場所が変更されています。(以下で、<Username>は、ログインユーザー名を意味しています)
ADMET Predictor™ 7.2 :
ADMET Predictor™ 8.0 :
MedChem Studio™のほとんどの機能が、MedChem Studio™ モジュールとして、
ケムインフォマティクス機能がいくつか新たに利用できるようになりました。これらの機能に対しては、追加ライセンス購入の必要はありません。
ユーザーインターフェイスが全面的に再設計され、データの可視化や操作がより容易になりました。
以上
注意:【Modeler モジュールをお持ちのお客様】前回のリリースであるバージョン7.0において、ディスクリプターの追加や更新が行われています。6.5以前のバージョンからアップデートされる場合は、7.2を使用される前に、構築したモデルで再学習させてください。
以上
注意:【Modeler モジュールをお持ちのお客様】前回のリリースであるバージョン7.0において、ディスクリプターの追加や更新が行われています。6.5以前のバージョンからアップデートされる場合は、7.1を使用される前に、構築したモデルで再学習させてください。
ADMET Predictor用のKNIME Workflowノードが追加されました。
Metabolism Converterの不具合が修正されました。
SDやRDファイルに保存する際に予測結果とS+pKaのマイクロステート情報を一緒に、あるいは別々に保存することができるようになりました。
ライセンスに関係するプログラムの不具合、およびマニュアル、ツールチップにおける単位表記の誤りが修正されました。
SaveメニューのSDF及びRDFオプションが修正されました。
Descriptor Sensitivity Analysis画面において、範囲外のディスクリプターを容易に識別したり、ソートしたりできるように、名前の先頭に**が追加されるようになりました。
出力フォーマット(DAT, SDF, RDF)を指定するオプション'-u'が追加されました。
S+pKaのマイクロステート情報を書き出すオプション'-z'および'-w'が修正されました。
ディスクリプターの相関フィルターとTLAによるディスクリプターの選択がより効率化するように改良されました。
いくつかの不具合が修正されました。
以上
注意:ディスクリプターが追加・更新されていますので、ver6.5以前で構築したモデルは、新しいバージョンで再学習させる必要があります。
pKa予測モデル(S+pKa)のデータセットの数を増やし、主に製薬企業に特有の化学構造領域の方向へケミカルスペースが拡張されました。詳細は、2) モデル を参照ください。
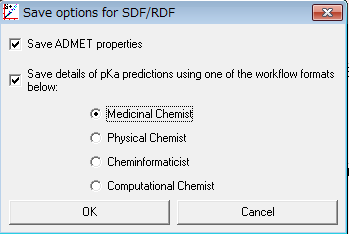
SDFフォーマットの出力オプションが改善されました。医薬品化学、物理化学、情報化学、コンピュータ化学といった各々のワークフロー特有のニーズに適合するようなS+pKaのパラメータをインタラクティブモードおよびコマンドラインモードで保存できるようになりました。5. コマンドラインを参照ください。
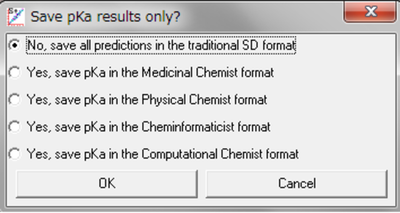
有機分子の特定イオン性官能基のイオン化挙動を抽出するためにデザインされた、新しいコンセプトを実装することで、pKa予測機能が強化されました。これには、Averaged Single Proton Acidity (ASPA)の斬新なコンセプトと同様にタンパク質化学者に知られているAveraged Site Protonation (ASP)とSingle Proton Midpoint (pK50)が含まれます。4. GUIを参照ください。
Artificial Neural Network Ensembles を基にしたバイナリ分類(ANNC)モデルの予測信頼性を定量化する新しい統計理論がADMET Modelerの自動機能として組み込まれました。6. ADMET Modelerを参照ください。
最適モデル選択のための、新規で有効なアルゴリズムが追加されました。6. ADMET Modelerを参照ください。
代表的な5つのヒトチトクロームP450(1A2、2C9、2C19、2D6、3A4)の

GastroPlus™ Drug Tables へのエクスポート機能が再編されました。Drug Tableに次の7つのカラムが追加されました。:血液・プラズマ濃度比、SGF、FaSSIF、FeSSIFの溶解度、角膜透過性および分子半径の2つの大きさ(長径・短径)。Drug TableとADMET Predictorスプレッドシートカラムとの間の割り当てをプログラムの外に出し、“GastroPlusTables.inp”という新しいサポートファイルを通してアクセスできるようになりました。
ADMET Risk計算のアルゴリズムが、”ソフトな(幅を持たせた)”決定境界に対応するようになりました。
ADMET Predictorと他のSimulations Plusのプログラムとの通信が大幅に再編され、改善されました。
MedChem StudioとMedChem Designerに提供しているADMET Predictorの計算エンジンが64ビット版にネイティブ対応されました。
“Esc”キーにより、計算途中で一時停止や停止ができるようになりました。
新規予測モデル
SimplePeff: ヒト空腸透過性の単純線形モデル、デフォルトではOffになっています。
S+pKaモデルが、大幅に見直されました。バイエル社から提供された高品質な19,464のpKa実測値が加えられ、これまでのpKa実測値14,176と合わせ、33,640の実測値からモデルが構築されました。新しいpKaモデルは、モデルの学習に使っていない、大規模な実測値3セットを用いてバイエル社で検証されました。以前のpKaモデル(パブリックドメインのpKaのみで組んだモデル ver6.5)との比較テストでは、二乗平均平方根誤差(RMSE)が50%も減少した例もありました。具体的には、テスト1(バイエル社の4,730化合物)のRMSEは、0.82(v6.5)から0.41 (v7.0)、テスト2(バイエル社の8,931化合物)のRMSEは、0.79 (v6.5)から0.52 (v7.0)、テスト3(バイエル社の12,951化合物)のRMSEは、0.72 (v6.5)から0.5 (v7.0)と減少しました。
新規ディスクリプターが追加されたため、全てのモデルが再構築されました。
S+PgpモデルがS+Pgp_Substrと改名されました。
Km、VmaxおよびCLint値を予測する、代謝のCYP反応速度(METAR)モデルは、CLint = Vmax/Km比で、互いに従属するようになりました。加えて、異なる発現レベルは、別の単位で表現されるようになりました。例えば、原子レベルのVmax値は、nmol/min/(nmol enzyme)で表現され、一方、分子レベルの平均Vmax値は、nmol/min/(mg microsomal protein)で表現されます。ミクロゾームでのCYP表現は、編集可能となり、ユーザーが望むように変更できるようになりました。
選択されたバイナリ分類モデルで予測信頼性が表示されるようになりました。6)ADMET Modeler を参照ください。
新規ディスクリプター
Rads_3D:分子平均包括配向が投影された幾何学的半径
T_Rads:Rads_3Dの2Dトポロジー版
ディスクリプターのアップデート
(カスタマイズされたHuckel法で計算された)pi サブシステムの部分原子電荷
分子構造の描画が更新されました。
互変異性体の描画で、プロトン移動を示すカラー表示が加えられました。
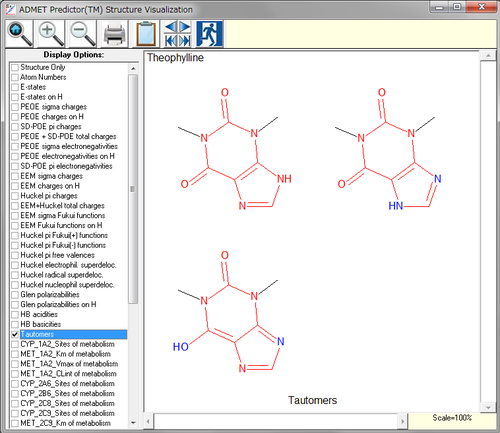
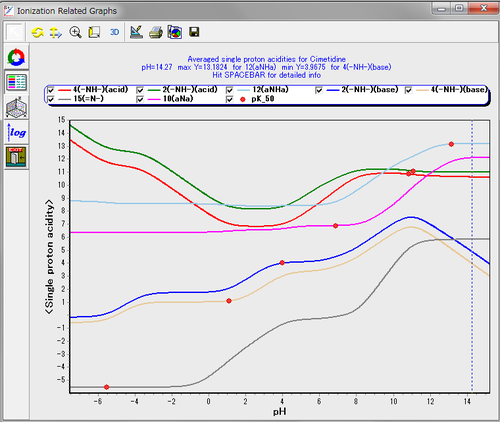
ASPA:Averaged Single Proton Acidity(平均シングルプロトン酸性度)。特定のpHおける個々のイオン基のマイクロステート平均有効”pKa” - pHの関数としての平均酸性度/塩基性度 ”強度”となります。
ASP:Averaged Site Protonation(平均部分プロトン化)。各pHで計算された個々のイオン性基の平均部分プロトン化で、0(非プロトン化)から1(プロトン化)で連続して変化します。ASPは、マルチプロトン性分子にある所定の基の”滴定”プロファイルとして解釈されます。
pK50:Single Proton Midpoint(シングルプロトン中間点)。ASP曲線が0.5に等しい点でのpH値に対応しています。すなわち、興味の対象となるイオン性基の半分がプロトン化した点です。環境の影響(他のイオン性基)を取り除かれた後の、マルチプロトン化分子における、基固有の酸性度/塩基性度の最良の見積となります。
上記のプロットは、Protonation Microstates ウィンドウから起動される解析グラフで利用できます。加えて、SDFのアウトプットのうちMedicinal Chemist formatには、pK50の値が保存されます。
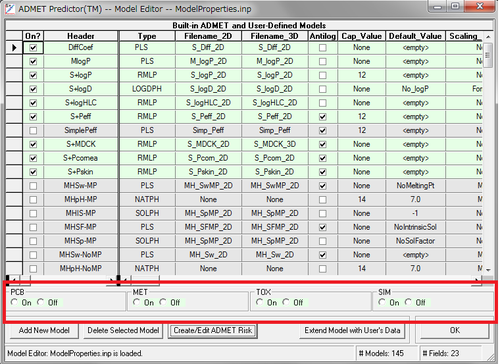
Model Editorに新しい機能を追加しました。
モジュールのライセンスに自動的に同期します。
モジュール全体のオン/オフが迅速にできるモジュールトグルボタンが加わりました。
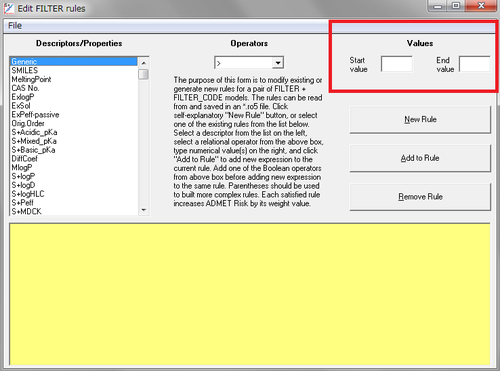
ADMET Risk Editorに“ソフトな”決定境界の入力が加わりました。
SDFアウトプットワークフローを調整するオプションである、-w と-s が追加されました。
GastroPlus™のtableエクスポート用のオプション、-g が追加されました。
特定モジュールのアウトプットを選択できる、-m が追加されました。このオプションを使うことによって、ディスクリプターカラムを減らすこともできます。
Artificial Neural Network Ensemblesをベースとした分類モデルに自動信頼性分析(Automatic Confidence Analysis- CFA)が組み込まれました。ANNCモデルの学習時にEnsemble Statistics Matrixの各々のセル内でCFAが自動的に稼働します。しかしながら、学習データ量と実測誤差率どちらかが非常に低い場合は、CFAが機能しない条件付稼働となります。CFAで学習させたモデルでは、信頼性の数値として、理論的に最低限の50%から上限100%までの値が、各々の予測に付加されます。
信頼性分析プロットである、“Wenkel plots”は、次のことを示します。
最適モデル選択機能(学習終了後のModel Matrix)が、新規で有効性の高い手法になりました。モデル選択の方法として斬新な統計法をベースにしました。最適モデルは、新しい色の体系で順位付けされます。
パフォーマンスグラフでのX、Y軸のスケールが同じになりました。
異なるアンサンブルを並べて比較できるようになりました。
利便性のため、Ensemble Statisticタブに”Stop”ボタンが加えられました。
以上
以上
以上
・S+logPおよびS+logDモデルを再構築されました。
・肝臓でのOATP1B1トランスポーターの阻害モデルS+OATP1B1を追加しました(新規モデル)。
・既存のisoform (1A2, 2C9, 2C19, 2D6, 3A4)の基質モデルを再構築されました。
・新規isoform (2A6, 2B6, 2C8, 2E1)の基質モデルが追加されました。
・新規isoform (2A6, 2B6, 2C8, 2E1)のSiteモデルが追加されました。
・Radb_3D: a geometrical version of the second ellipsoidal radius
・Radc_3D : a geometrical version of the third ellipsoidal radius
・HydroR_3D : a geometrical version of the hydrodynamic radius
・T_Rada : a topological version of the first ellipsoidal radius
・T_Radb : a topological version of the second ellipsoidal radius
・T_Radc : a topological version of the third ellipsoidal radius
・T_HydroR : a topological version of the hydrodynamic radius
・T_MIRxx: a topological version of the largest principal moment of inertia
・T_MIRyy: a topological version of the second largest principal moment of inertia
・メニューの「Recalculate」が「Calculate」に変更されました。
・メニューのADMET Modelerの下にあった「Descriptor Sensitivity Analysis」を「Calculate」の下に移動しました。
・構造編集ツールとして「MedChem Designer」が使用可能になりました。
・画面上でSMILESの編集が可能でしたが、使用不可となりました。これは、誤って編集されることも多いためで、構造編集はMedChem Designerで行うようになりました。
・メニューに「Edit」を加えました。この「Edit」で、Copy, Paste, FindおよびEdit Structureができるようになります。
・Molecular Spreadsheetのカラムの表示を選択できるようにナビゲーションボタンが追加されました。
・メニューに「View」が増えました。この「View」からカラム選択ができます。
・ナビゲーションボタン、アイコンの追加、およびマウスホイールでMicrostateや構造表示ができるようになりました。
・メニュー に「Tools」が加わり、以前の「Option」および「ADMET Modeler(TM)」をこの「Tools」の下に移動しました。
・CYPによる代謝部位表示が改善されました。
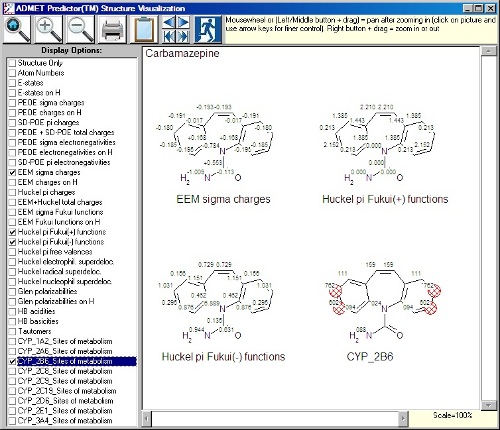
・Structure Visualizationウィンドウでの原子物性表記を下記のように複数の同時表示が可能になりました(CYP部位および互変異体含む)。
・ Miner3Dの操作が変更されました。
・ 計算の進捗表示のバーが表示されるようになりました。
・ 稼働環境として解像度を1024x768になりました。
・ Microstate表示からfaux両性イオンを削除されました。
・ Arithmetic operationに平方根が追加されました。
・ ModelOptionsウィンドウの中にあるADMET Risk editorが別ウィンドウになりました。
・ ANNEモデル設定オプションで偏向分類ANNEを選択できるようになりました。
・ 統計用コードをVBからADMETModeler.dllに変更されました。
以上
MDL Metaboliteあるいは、Symyx Metaboliteとして知られ、現在は、Accelrys Metabolite DatabaseとなっているDatabaseから10種の新しい予測モデルを構築しました(詳細は、Modelsに記載)。これは、別モジュールとなります。
オプションの -r をcommand line modeから削除しました(ADMET Risk filterのデフォルト)。
サーバとクライアントのPipeline Pilotコンポネントを修正し、Pipeline Pilot v7.5とv8に対応しました。
Simulations Plus社の新しい構造描画ツール「MedChem Designer™」とのリンクが可能となりました。
代表的な5つのCYP(1A2, 2C19, 2C9, 2D6, 3A4)での
|
|
|
|
|
|
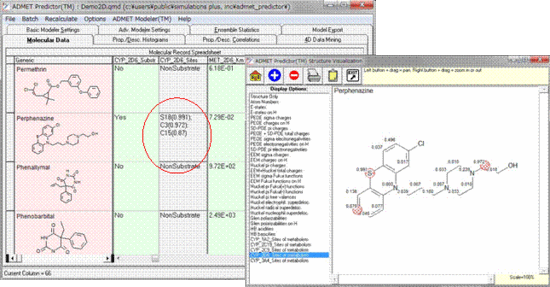
MET_2C19_Inh : CYP2C19に対しての阻害特性
ADMETという名称ながら、吸収(A)の部分しか評価していませんでしたので、ADMET_RiskとADMET_Codeの名称をS+Deflt_RiskとS+Deflt_Codeと変更しました。また、今後はすべてのADMETに関係するフィルターをADMET Riskと呼びます。
ADMET RiskフィルターがModelProperties.inpデータベースに存在する際に予測モデルとして扱われます。したがって、フレキシビリティーが増し、一つだけでなく複数のADMET Riskカラムを発生させることができます。WDI(World Drug Index)データベースを用いて、リピンスキーらと同じようなアプローチで、WDIの90パーセントの外に予測物性がある場合、アラートを出す新しいADMET Riskフィルターを構築しました。
新規ADMET Riskフィルターとしては、
Physicochemical & Biopharmaceutical Moduleが必要となります。
Physicochemical & Biopharmaceutical Moduleが必要となります。
Emslein Metabolism Moduleが必要となります。
基質ルールを含むことでより正確となっています。Enslein Metabolism ModuleおよびMetabolite Moduleが必要となります。
Moduleが必要となります。
リスクスコアです。全Moduleをお持ちであればすべてのリスクポテンシャルが見積もられますが、お持ちでないModuleがある場合は、その部分は評価されません。
S+Peffモデル構築の際、一つの化合物の構造に誤りがありましたので、修正しました。
処理速度を上げるためコンパクトな形式に変換されました。
変数をコントロールする、Cap_Value, Default_Value, Scaling_DescriptorおよびDetermining_Descriptorの機能を向上させました。文字列変数を割り当て、Scaling_Descriptorは、条件モデルを実行するトリガーとして使われます。
求電子、ラジカルおよび求核反応度のためのFukui`s superdelocalizability。
|
|
デフォルトのGasteiger`s pi electronegativityを変更しました。
以上
64bitのOS上で起動できるようになりました。(64bit用にチューニングはされていません。)
インストールプロセスを改善いたしました。
4.0では、ユーザの使用状況をログに残す(ADMET_Predictor_Usage.txt)ためには、Options->Usage logging でチェックしなくてはなりませんでしたが、5.0よりデフォルトでチェックが入るようになりました。
File/Openのメニューを簡略化しました。Fileタイプをファイル名の拡張子から自動的に認識するようにしました。
SMILESは、QMDファイルのみ対応しておりましたが、SMIフォーマットにも対応するようになりました。
TOX_SKIN -アレルギー性皮膚感作(マウス)
TOX_BCF - 環境中での生物濃縮係数
TOX_RAT - 急性致死毒性(ラット)
S+FaSSGF – 人工胃液での溶解度
S+FaSSIF - 人工腸液(食餌後)での溶解度
S+FeSSIF - 人工腸液(絶食時)での溶解度
S+Pcorneal - ヒト角膜透過係数
pKa予測モデルを見直しました。モデル作成時のデータベースに1,800以上の新規化合物を加えました。(2,400以上の新規実測pKa値を加え、モデリングデータベースサイズとしては13,881)
誤ったpKaアサイメントを修正しました。これらの改善によりpKaモデルは、トレニングセット、外部テストセットともRMSEが0.6以下となりました。
この改善により、pKaは、S+Acidic_pKa、S+Mized_pKa、S+Basic_pKaの3つのカラムに表示されます。
S+社で開発した社内用のデータキュレーション用ツールDirectory of Moleculesにより重複する構造は排除し、再構築し、予測精度を高めました
a) カラムのマージ
b) 複数のpKaに対応するRefAllPKaを追加しました。
数百の新しいトレーニングサンプルで部分原子電荷およびFukui指数のモデル性能を向上させました。
予測モデルがより良くなるようFukui functionの異なるスケーリングを導入しました。
医薬品でホウ素含有化合物が多く存在するため、パラメータを最適化し、有機エレメントに追加しました。
新しいDescriptorは、N_Boron, SsssB, SaasBとなります。
非常に特殊な配列に存在する –NH-原子を追加しました。例えば、A,B=C,NのZ(Y)A=B-NH-、Z,Y=-X(=O)- や -C#N、O=X-OH以外のX=C,S,N
スルフォンアミドのNをリストから削除しました。
先端ab initioの結果から超原子価のSやPを含む環を芳香族として認識しなくなりました。
カルボニウムおよび芳香族スルフォニウムを追加しました。
四価のNとPを取扱えるようになりました。水素は原子価に追加されませんが変わりにNとPは+1の形式電荷を受取るようになります。
以前のNitroso descriptorは最適なニトロソ基として制限され、一方、N-オキサイドは、新しい「N-オキサイド_>[N+][O-]の中に存在しています。イオン化N-オキサイドの定義を広げました。
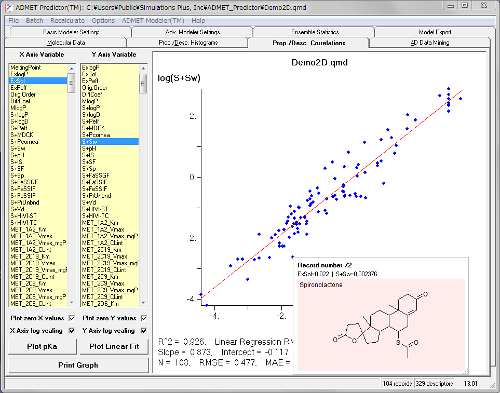
Prop./Desc. Correlationsタブに”Plot pKa”ボタンが追加されました。実測pKa値と予測pKa値の相関がわかるようになりました。
Prop./Desc. Correlationsタブのグラフのポイントをクリックすると構造が表示されるようになりました。
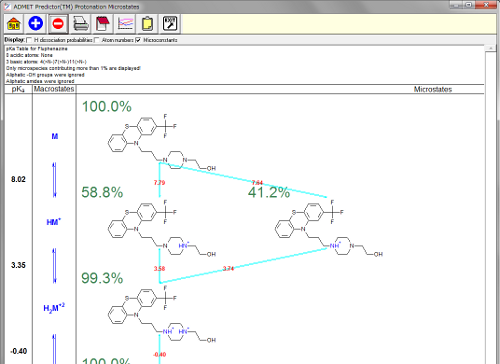
Molecular Spreadsheetの行のpKaをクリックするとpKaミクロステートの図が変更されます。また構造をクリックするとStructure Visulalizationがクリックした構造に変更されます。(4.0までは一度Exitしないと次の図が表示されませんでした。)
ディスプレイオプションのリストにマイクロコンスタントが追加されました。デフォルトの最小のマイクロステートの貢献度が10%から1%に変更されました。
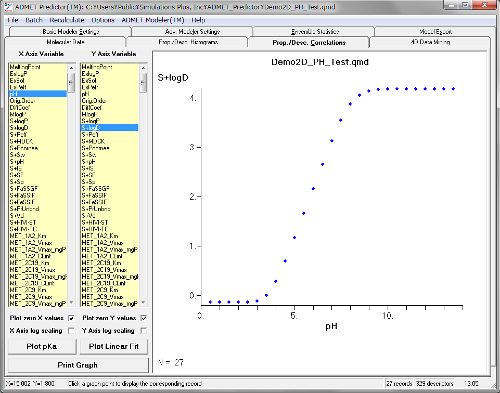
Run OptionでpHカラムをサポートするようになりました(化合物ごとのカスタムpH値)
構造ファイルに化合物ごとのpH、または同じ化合物でpHの数値をいくつも入れることで化合物ごとのpHでの計算を行います。下の例は、同じ化合物を細かくpHをとって溶解度を計算した例です。
不均衡データセットの取扱の先進的な方法および予測モデル評価の新しい統計手法を追加しました(Specificity,Sensitivity, Youden index, Matthews Correlation Coefficient)
バイナリANN分類モデル用のDescriptor Densitivity Analysisの手法を開発しました。
Single threshold optimization methodを加えました。
k-meansアルゴリズムを基にしたTest set selectionを加えました。
k-means サブセッティングを導入することで、Genetic Algorithmの適応を広げ大規模なデータセットに対応できるようにしました。
Model graphing window上のポイントをクリックするとそのポイントの構造が表記されます。

全てのTest selection methodsを可能にし、Model retraining optionを拡張しました。
Ensemble Statisticsタブのtest statisticsおよびverification statisticsの色(青/赤)を変更しました。
以上
注意:このバージョンでは、ディスクリプターが変更および追加されています。オリジナルのモデルを構築されている場合、モデルパラメータは新しいディスクリプターの値と互換性がありませんのでオリジナルモデルで計算されません。新しいディスクリプターで再構築されることを強く推奨します。今回のversion4.0では、再構築を比較的短時間で行える新しい機能「Model Retraining」が追加されていますのでご利用ください。
目的とする血中薬物濃度になるような最適な薬物投与量を予測する「SimDOSE」モデルが追加されました。SimDOSEモデルは新しいオプションシミュレーションモデルでSimHIA(simulated fraction absorbed)と共に用います。SimHIA、SimDOSEとも新しいModule(Simulation Module)となり、ADMET PredictorのModuleは、5つとなります。
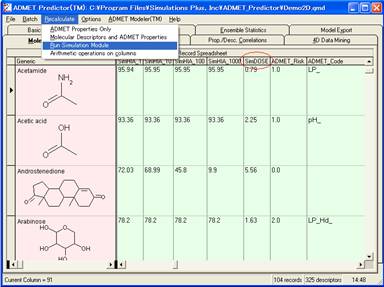
Simulation Moduleで使用する、.hiaファイルフォーマットがよりフレキシブルになりました。今までは、.hiaファイルフォーマットは、キーワードとパラメータの位置が固定でしたが、今回のバージョンよりパラメータの数や種類を変更することが可能となりました。
Simulation Moduleに新しいODEインテグレータである、CVODEを使用し、GastroPlusとの互換性を高めました。
すべての消化管シミュレーションモデルが、コマンドラインから利用できるようになりました。計算結果は、タブ区切りのフォーマットである、“.osm”(Output of Simulation Module)というファイルになります。このファイルをPipeline Pilotへ流し込むことも可能です。タブ区切りのファイル(.dat、.out、.txt)は、コマンドラインモードでの入力として利用することができるようになりました。このようなファイルのインプットは、
pH依存性水-オクタノール分配係数であるLogDの予測にユニークなモデルを追加しました。今までのS+logDは、n-オクタノールへのイオン化種の分配の予測に一定の修正率を用いていました。今回は、予測精度を大幅に上げるために人工ニューラルネットワークアンサンブルを用いて分子構造からそれらのファクターを予測します。
ヒトの肝臓に悪影響を及ぼす薬物の予測モデルをFDAのデータベースから構築しました。アルカリホスフォターゼ(ALP)、SGOT、SGPT、LDH、GGTといった肝臓の酵素の診断で数値上昇に大きく影響を与える分子の分類を行います。
Uridine 5`-Diphosphate-Glucuronosyltransferase(UGT)の7つのイソ型である、1A1、1A3、1A4、1A6、1A9、1A10、2B7によるグルクロン酸化の予測モデルをEnslein Instituteのデータから構築しました。

複数ライセンス(ネットワークライセンス)をお持ちの場合、複数台のPCで同時にライセンス数の起動は可能でしたが、1台のPCで複数のADMET Predictorを起動できませんでした。今回、複数ライセンスをお持ちの場合、1台のPCで複数のADMET Predictorを起動させることが可能となりました。
Miner 3D™のバージョンが、最新版である7.2.6と更新されました。
Miner 3D™インターフェースでフルデータマトリックスを利用可能にすることにより、データカラムのハンドリングが格段に向上しました。また、前のバージョンでは、+ や - 、_、といった特殊なキャラクターが表示できませんでしたが、今回から表示できるようになりました。
エラーログファイルは、ADMET Predictorのフォルダーに保存されていましたが、input fileが置いてあるフォルダーに保存されるようになりました。
logDとpHの機能としての溶解度プロファイルの計算を取扱うサブルーチンを見直ししました。
まず、ゼロ荷電状態ですべての種をカバーするように関連する塩基の量である、logPと固有溶解度の定義を更新しました。さらに微視的な観点よりむしろ巨視的な観点で酸と塩基の解離を定義しました。その結果、logDとpH依存性溶解度の変数は、単純化されて改善されました。両性イオンの固有溶解度と溶解度ファクターの計算の値で特に顕著な影響があります。
GastroPlus™ Acid/Base TableへのSaveオプションにGastroPlus™とDDDPlus™のpKa tableへのインポート用として新たにいくつかのカラムを追加しました(NumAcidGroups、FormalCharge、FractionZwitterionic、PAPN、PCPN、PZPN - 最後の3つのパラメータは、新しいlogDモデルの構造パラメータです)。これらのパラメータは、新しいlogDと溶解度で用いられます。
モデリングでのN_Kekuleの置き換えのために作られました。
原子の数で割った距離行列の最大要素
P. Ertlらによって定義されました。
原則的には、古いディスクリプターですが、新しい定義に基づいて計算されています。FUnionは、総電荷ゼロでのすべての種のpH依存部分を表しています。FZwitterは、両性イオンの状態で存在するFUnion種のpH独立成分を表しています。
構造表示のみ
未知のE-typeの場合でのEEMの電気陰性度と硬度のデフォルト値を0でない値としました。
配位のE-Typeの割当てと荷電状態を更新しました。
pKaが無い場合、”None”と表示されるようになりました。
前のバージョンで構築したモデルの再トレーニングを容易にする機能を追加しました。
新しいデータやマイナーチェンジによりモデル再構築を行う際に時間と労力を節約できます。毎回、モデルを始めから作り直す代わりにディスクリプター選択とモデルの最適化に対して、既存のモデルを最小限の労力で再最適化できます。
モデル構築後にSALI (Structure-Activity Landscape Index)の曲線を計算し、表示するオプションが追加されました。R. GuhaとJ.H.Van Drieによって構築された、SALIおよび関連するSCI (SALI curve integral)のコンセプトは、in silicoモデルによってMaggioraの”activity cliffs”を捉えるためのものです。ADMET Modelerのメニューから選択できます。
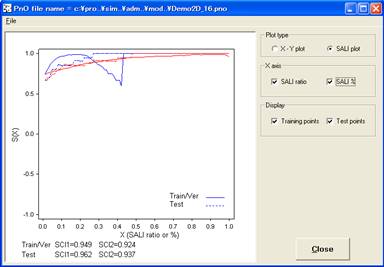
以上
全く新しい部分電荷モデルが実装されました。これはハイレベルな量子理論で計算したab initio波動関数のNatural Population Analysis法に基づくものです。
3重結合は2つの垂直方向のπ共役系(pz及びpy)として取り扱うようになりました。
このことによりπ電子の電荷を正しく計算することができようになり、またクムレン化合物を適切に取り扱うことができるようになりました。
構成上のディスクリプター1種、トポロジカルディスクリプター2種、原子荷電ディスクリプター16種、原子反応ディスクリプター36種追加しました。
以下の置換基の配位結合は2重結合としてではなく電荷で表現されるようになりました。
(Nitro, NNitro, N-oxide, Nitrosamine, Azoxy, Azide, Nitrate, Oxadiazooxide, Nitrosohydroxyamine, aromatic Sulfoxide)
ディスクリプターの変更・追加によりADMET Riskを含むすべての予測モデルを再構築した結果、version2.4.0と比較して、同様あるいはそれ以上の精度となりました。
GastroPlusの最新versionに合わせて小腸内の水分量を変更した系での吸収率に変更しました。
S+logDモデルのパラメータを更新したことで、予測精度が向上しました。
S+Vdモデルをヒト定常分布容積の実験値の大規模なデータベースで再構築しました。
14の新しい予測モデルを追加しました。その結果、予測モデルは全Moduleで77種類となりました。
モデル構築時のディスクリプター選択のためのアルゴリズムを2種新規追加しました。
旧versionでは、テストセット選択のnon-Kohonen法でtrain:verifyが1:1でしたが、今回は、Kohonen法に合わせて2:1に変更しました。
多相関ディスクリプターをrandomly (デフォルト)、またはTLA rankが最も低い値によって排除できるようになりました。
File pathの記載を取り除きシンプルなファイル名のみの表記としました。
Dataの視覚化をよりよくするためにMiner 3DグラフィックModuleを追加しました。
アトムナンバーおよび全ての脱プロトン化構造を表示できるようになりました。
21タイプの原子特性(チャージ、分極率など)が表示できるようになりました。
インディケーターが表示されるようになりました。
右クリックでCopy, Paste, Findができる機能が加わりました。
アウトレイヤーインディケータの色を赤からマジェンタに変更しました。
計算結果のグリッド表示が自動的に保存されます。また、右クリックでデータをクリップボードに保存することができるようになり、
タブ画面をBMP Fileで保存できるようにもなりました。さらにセルをクリックするだけで各Modelのperformanceを表示できるようになりました。
以上
多くの既存のチャージに関連するディスクリプターは、推定NPAチャージに切り替わりました。また、いくつかの分子ディスクリプターでは原子順位依存に対しての影響を排除しました。
再構築により、version 2.0.1と比較して同等以上の値となりました。
非局在化piシステムでのチオン基の新しいpKaモデルを追加しました。
GastroPlusのOptimizationモジュールで構築されたモデルです。
サルモネラ菌10種の菌株でのAmesの変異原性試験の新モデル(TOX_MUT_*)を予測モデルに加えました。
HIV-1 インテグラーゼ 3'-processing阻害とHIV-1 インテグラーゼstrand transfer阻害の予測モデルを追加しました。
TOX_BRM_Sal、S+_S*_Drugsと古いS_Sw_*溶解度モデルが削除されました。
インタラクティブモードで行うほぼ全ての計算がコマンドラインで実行できるようになりました。
2D SDFや2D RDFでも保存できるようになりました。
モデルの選択や読込みが簡易になり、3D表示もできるようになりました。
Atom Numberを表示することもできます。
既存のモデル式に誤って上書きするとメッセージが表示されます。
ログは、ADMET_Predictor_Errors.logに記録されます。
pKaのセルをクリックすることで詳細を表示するようになりました。
新たにBase-10 exponetial functionが加わりました。
paired-t-testと呼ばれる新しい手法が加わりました。
Yes/Noや0/1やActive/Inactiveなどのロジスティック関数も対応可能となりました。
これによりSVMモデルファイルのサイズが大幅に縮小しました。
ANNやSVMのような非線形モデルではAICよりQ^2の方が適しているからです。
エクスポートするアンサンブルモデルのサブセットを選択できるようになりました。
モデル構築のためのテストセット選択の新しい手法となります。
独自アルゴリズムにより多相関ディスクリプターを削除するようになりました。
トレーニングポイントやテストポイントの表示をオンオフできるようになりました。
以上
ADMET Modelerで作成したモデル式をADMET Predictorにシームレスに追加できます。ADMET PredictorとADMET Modelerは別プログラムということで各々の価格が付いていましたが、Version2.0よりADMET Predictorの価格にて両プログラムをお使いいただけるようになりました。
ログを見ることで現在使用しているユーザを知ることができます。。
J-Alertを「ADMET_Risk」にJ-Codeを「ADMET_Code」に名称を変更しました。さらにADMET_Riskの個々のルールに重み付けができるようになりました。
自動ディスクリプター選択機能が「Column Selection」ウィンドウで操作できるようになりました。
Molecular spreadsheetのカラム間の数値の加減乗除、対数、逆対数を計算することができます。
classification support vector machines (CSVM) とkernel partial least squares (KPLS)の2つの新しいモデルを認識するようになりました。
一般的なタブ区切りのファイルも読込むことができるようになりました。全てのTab画面で拡大縮小が可能となりました。FileをADMET Predictor上で編集できるようになりました。Correlation graphイメージとデータを保存してWindowsクリップボードにコピーすることができるようになりました。
以上
©2004-2025 Northern Science Consulting Inc. All Rights Reserved.


 ADMET Predictor 製品資料
ADMET Predictor 製品資料